Construction and Validation of A Prognostic Model of Lung Adenocarcinoma Based on m5C Modification-Related Genes
-
摘要:目的
基于m5C修饰相关基因构建并验证肺腺癌(LUAD)预后模型,并探讨其临床应用价值。
方法基于TCGA和GSE30219、GSE31210、GSE50081等LUAD数据集资料,通过单因素Cox、Lasso、多因素Cox回归分析筛选预后相关基因并构建模型,通过绘制Kaplan-Meier曲线、ROC曲线及Cox回归观察模型的稳健性和预后性能,并探讨模型与临床病理特征的相关性。
结果构建了由CDK1、CDKN1A、NOP2、RRM2、TCL6、TLR8、TRDMT1、YTHDF2等8个m5C修饰相关基因组成的预后模型。风险评分是LUAD患者预后的独立危险因素,其联合T分期和N分期的列线图能更准确地预测患者的预后。高风险组患者中巨噬细胞、CD4+/CD8+T细胞等浸润显著减少。LUAD组织中风险评分显著高于正常组织,且与T分期、N分期正相关。吸烟、EGFR野生型患者的风险评分显著升高。
结论基于m5C修饰相关基因构建的预后模型,对预测LUAD患者的预后评估具有较好的准确性和稳定性,且与患者的临床特征、驱动基因突变、免疫浸润等密切相关,可为LUAD的治疗与预后评估提供潜在依据。
Abstract:ObjectiveTo construct a prognostic model of lung adenocarcinoma (LUAD) based on m5C modification-related genes and to explore its clinical value.
MethodsBased on the LUAD data in TCGA, GSE30219, GSE31210, and GSE50081 cohorts, prognosis-related m5C modification-related genes were screened, and the prognostic model was constructed by using univariate Cox, Lasso, and multivariate Cox regression analyses. Kaplan-Meier curve, ROC curve, and Cox regression were used to observe the robustness and prognostic performance of the model. The correlation between the prognostic model and clinicopathologic features was further explored.
ResultsA prognostic model consisting of eight m5C modification-related genes, including CDK1, CDKN1A, NOP2, RRM2, TCL6, TLR8, TRDMT1, and YTHDF2, was constructed. Risk score was an independent risk factor for the prognosis of patients with LUAD, and it is combined with age, T stage, and N stage to constitute a nomogram which can accurately predict the prognosis of patients. The infiltration of macrophages and CD4+/CD8+T cells was significantly reduced in high-risk patients. The risk score in LUAD tissues was significantly higher than that in normal tissues and was positively correlated with T stage and N stage. The risk score of smoking and EGFR wild-type patients was higher than that of non-smoking and EGFR-mutant patients.
ConclusionThe prognostic model constructed based on m5C modification-related genes has shown good accuracy and stability in predicting the prognosis of patients with LUAD, and it is closely related to clinical features, driver gene mutations, and immune infiltration, which can provide a potential basis for the treatment and prognostic assessment of LUAD.
-
Key words:
- Lung adenocarcinoma /
- Methylation modification /
- m5C /
- Prognosis model /
- Risk score /
- Nomogram
-
0 引言
肺癌的发病率和死亡率在世界范围内居高不下,其中肺腺癌(Lung adenocarcinoma, LUAD)是最常见的病理类型,早期症状不明显,易被忽视,确诊时常处于晚期[1]。近年来,尽管LUAD的诊断技术和放化疗、免疫治疗、靶向治疗等治疗手段不断进步,但LUAD患者的5年生存率约为20%,预后不佳[2]。早期发现、及时干预、合理的预后评价是影响肺腺癌预后的关键因素。因此,探索影响LUAD患者预后的生物标志物,开发新的模型来更准确地预测患者的预后情况至关重要。
RNA存在100余种化学修饰,甲基化是主要的修饰形式,包括N1-甲基腺苷(N1-methyladenosine, m1A)、5-甲基胞嘧啶(5-methylcytosine, m5C)、N6-甲基腺苷(N6-methyladenosine, m6A)和7-甲基鸟苷(N7-methylguanosine, m7G)等[3]。来自供体的一个活性甲基被添加到RNA中胞嘧啶碱基的碳5位置以形成m5C修饰,是一种广泛存在的RNA修饰,在mRNA和非编码RNA中都有检测到,包括tRNA、rRNA、snRNA和eRNA等。m5C修饰主要由甲基转移酶、去甲基化酶和识别蛋白介导[4]。
tRNA中的m5C修饰主要参与维持稳态、优化密码子-反密码子配对、调节应激反应和控制翻译效率。mRNA中的m5C则与mRNA稳定、剪接和核质穿梭、DNA损伤修复、增殖和迁移、干细胞的发育、分化和重编程等多种生物学进程有关。mRNA m5C的异常修饰与多种疾病相关,包括动脉硬化、自身免疫性疾病和癌症等[5]。近年来,有报道称mRNA m5C的异常修饰参与膀胱癌、胃癌和食管鳞状细胞癌的发生发展过程[6-7]。目前m5C相关基因对LUAD预后影响的研究较少,本研究就此构建了LUAD的预后预测模型,评估了新模型的预后预测价值,同时进行了免疫浸润分析,以期为LUAD的临床治疗提供新的参考。
1 资料与方法
1.1 数据收集与整理
TCGA和GEO(GSE30219、GSE31210、GSE50081)数据库中LUAD患者的临床资料及转录谱数据获取同课题组前期研究[8]。其中TCGA、GSE30219、GSE31210、GSE50081数据库中分别包含LUAD样本535、124、226、129例,正常样本59、14、20、0例。在进行预后分析时,均剔除随访资料不完整、随访时间≤30天的样本。从人类基因数据库官网(https://www.genecards.org/)中检索并下载获取136个m5C修饰相关基因。
1.2 差异表达基因分析
采用DESeq2包对TCGA数据库中的数据进行差异表达基因分析,筛选标准为|log2(差异倍数)|>1且调整后P<0.05。
1.3 富集分析
通过基因本体功能(Gene ontology, GO)分析预后相关m5C修饰相关基因参与的生物过程(Biological process, BP)、细胞组分(Cellular component, CC)、分子功能(Molecular function, MF),通过京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)分析m5C修饰相关基因参与的主要信号通路。利用提供的分子数值通过GOplot包计算每个富集条目对应的z得分值,使用ggplot 2包对富集分析结果进行可视化。
1.4 预后模型的建立
参照本研究课题组之前建立的流程与方法[8],使用caret包对TCGA数据集按照7∶3的比例进行随机分层抽样,获得训练集(346例)和验证集(148例)。使用survminer包对训练集行单因素Cox回归分析,初步筛选预后相关的m5C修饰相关基因,为避免遗漏,设定P<0.2做进一步分析。随后使用glmnet包进行最小绝对收缩和选择算子(Least absolute shrinkage and selection operator, Lasso)回归分析缩小候选基因范围。最后通过多因素Cox回归分析建立预后模型。在TCGA全集、GSE30219、GSE31210和GSE50081中根据预后模型分别计算患者的风险评分:风险评分= $ {\displaystyle\sum }_{i=1}^{n}exp_i\mathrm{*}Coef_i $(其中exp和Coef分别代表DRGs的表达量和风险系数),并以各数据集中风险评分的中位值为界,将患者分为高风险组和低风险组。
1.5 预后模型的验证
预后模型的验证方法同李梦涵等[9]的研究。使用survival包对TCGA训练集、验证集及全集中LUAD患者进行Kaplan-Meier生存分析。使用timeROC包绘制ROC曲线,评估该模型对LUAD患者1、3、5 年生存期的预测效能。在GSE30219、GSE31210和GSE50081这3个测试集中重复上述操作进行外部验证。
1.6 列线图、校准曲线和决策曲线绘制
在TCGA数据集中,纳入年龄、性别、吸烟、T分期、N分期、肿瘤分期和风险评分进行多因素Cox回归分析。纳入LUAD患者独立的预后因素,使用rms包绘制列线图,同时绘制校准曲线用于评估实际生存率和预测生存率的一致性。使用dcurves包绘制决策曲线(Decision curve analysis, DCA),观察列线图模型预测患者预后的优越性。
1.7 免疫浸润分析
在TCGA数据集中,通过ESTIMATE算法评估高风险组和低风险组肿瘤纯度、基质评分、免疫评分和综合评分的差异,通过TIMER算法评估高风险组和低风险组6种免疫细胞丰度的差异。从文献[10]中收集常见免疫检查点,比较高风险组和低风险组两组间36种免疫检查点表达的差异。
1.8 统计学方法
采用R 4.3.1软件进行数据分析及绘图。两组间计量资料比较采用Wilcoxon秩和检验,多组间比较采用Kruskal-Wallis法。P<0.05为差异有统计学意义。
2 结果
2.1 肺腺癌特异性m5C修饰相关基因筛选
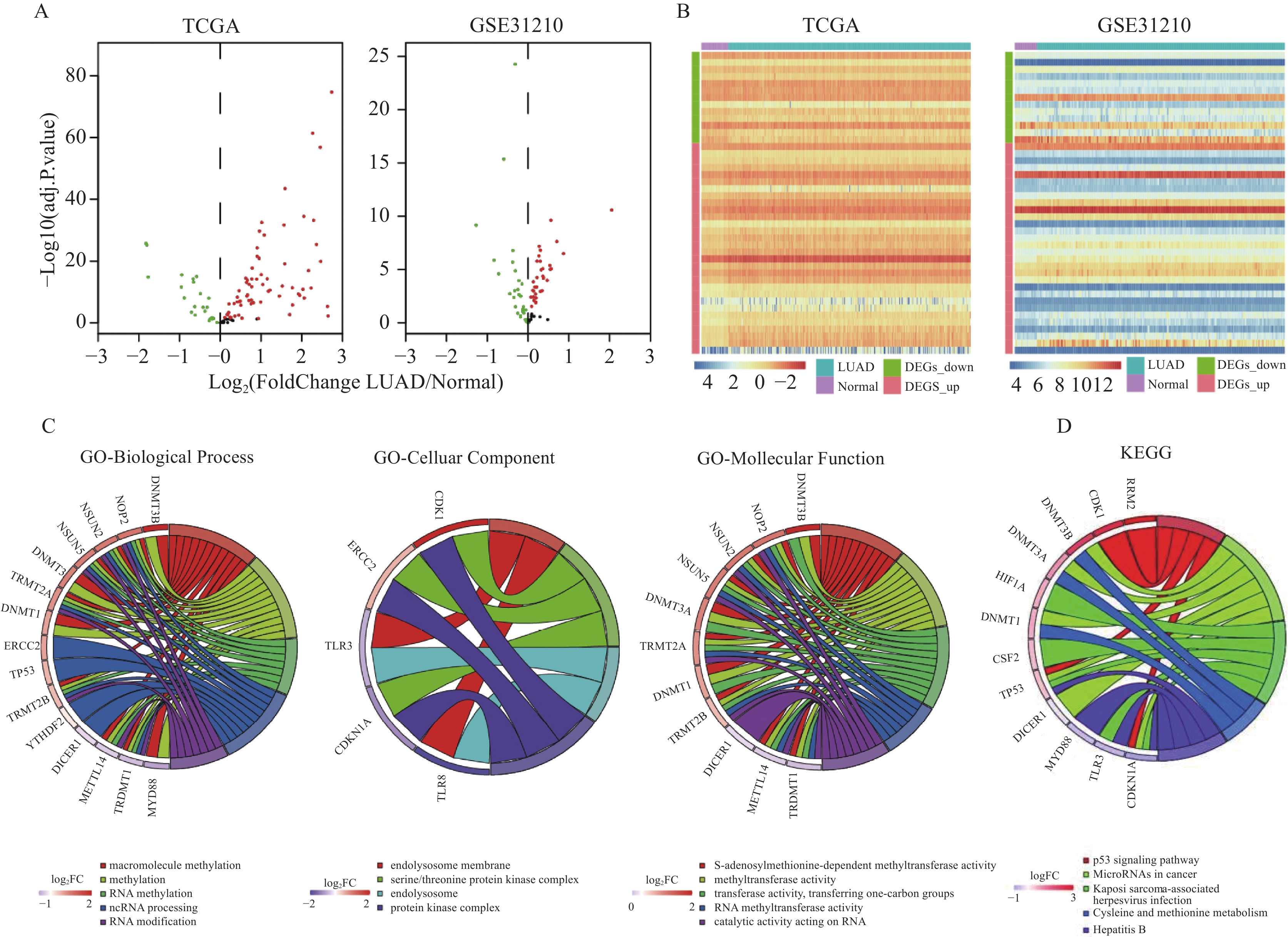
在TCGA数据库中筛选出9 878个基因在LUAD中表达上调,2 843个基因在LUAD中表达下调(P<0.05)。 在GSE31210数据集中筛选出329个基因在LUAD中表达上调,559个基因在LUAD中表达下调。将上述基因与136个m5C修饰相关基因取交集,在TCGA数据集中,共获得75个表达上调的m5C修饰相关基因和21个表达下调的m5C修饰相关基因,在GSE31210数据集中共获得34个表达上调的m5C修饰相关基因和18个表达下调的m5C修饰相关基因。两个数据集中共同表达上调的m5C修饰相关基因有30个,共同表达下调的有13个。
为进一步探索筛选出的差异化表达基因在LUAD中潜在的生物学功能和信号通路,我们进行了GO/KEGG分析。在生物过程方面,主要与高分子甲基化、甲基化、RNA甲基化、ncRNA加工、RNA修改等有关;在细胞组分方面,主要与内溶酶体膜、丝氨酸苏氨酸蛋白激酶复合物、内溶酶体、蛋白激酶复合物等有关;在分子功能方面,主要与s-腺苷蛋氨酸依赖的甲基转移酶活性、甲基转移酶活性、转移酶活性、转移单碳基团、RNA甲基转移酶活性、作用于RNA的催化活性等有关。同时也与p53信号通路、肿瘤中的microRNA、卡波西肉瘤相关疱疹病毒感染、半胱氨酸和蛋氨酸代谢、乙型肝炎等通路相关,见图1。
![]() 图 1 肺腺癌特异性表达m5C修饰相关基因的筛选Figure 1 Screening of specifically-expressed m5C modification-related genes in LUADA: volcano plots revealed differentially expressed genes between LUAD tissues and normal tissues in TCGA and GSE31210 cohorts; B: heatmap revealed the expression level of m5C modification-related genes in TCGA and GSE31210 cohorts; C, D: GO and KEGG enrichment analyses for m5C modification-related genes in TCGA cohort.
图 1 肺腺癌特异性表达m5C修饰相关基因的筛选Figure 1 Screening of specifically-expressed m5C modification-related genes in LUADA: volcano plots revealed differentially expressed genes between LUAD tissues and normal tissues in TCGA and GSE31210 cohorts; B: heatmap revealed the expression level of m5C modification-related genes in TCGA and GSE31210 cohorts; C, D: GO and KEGG enrichment analyses for m5C modification-related genes in TCGA cohort.2.2 基于m5C修饰相关基因的预后模型构建
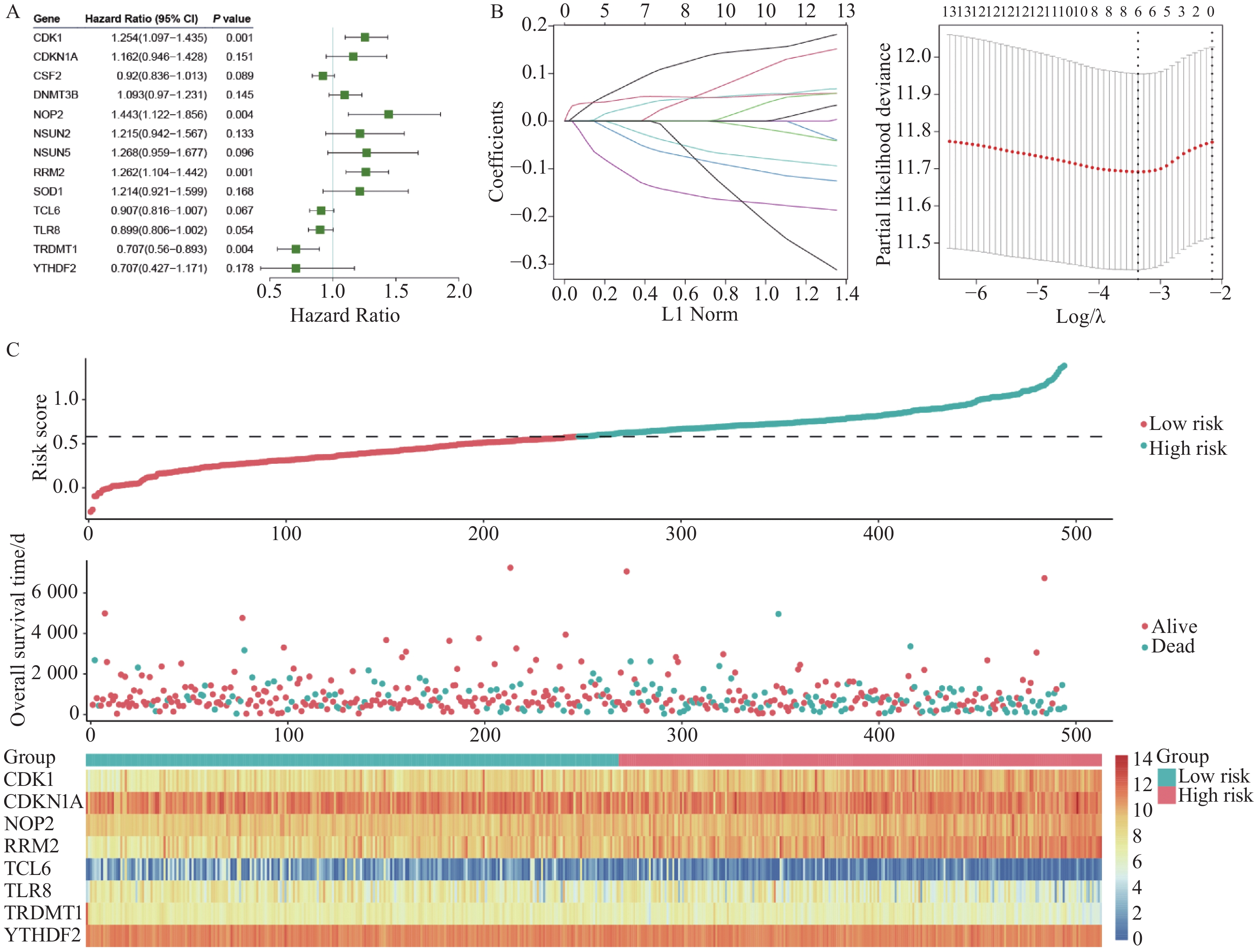
通过单因素Cox回归分析,初步筛选出13个m5C修饰预后相关基因,见图2A。然后通过Lasso回归分析,设制十折交叉验证获取最优模型,最终获得8个与肺腺癌预后独立相关的m5C修饰相关基因:细胞周期蛋白依赖性激酶 1(Cyclin-dependent kinase 1, CDK1)、细胞周期蛋白依赖性激酶抑制因子 1A(Cyclin-dependent kinase inhibitor 1A, CDKN1A)、核仁蛋白2(Nucleolar protein 2, NOP2)、核糖核苷酸还原酶调节亚基M2(Ribonucleotide reductase regulatory subunit M2, RRM2)、T细胞白血病/淋巴瘤基因6(T cell leukemia/lymphoma 6, TCL6)、Toll样受体8(Toll-like receptor 8, TLR8)、tRNA天冬氨酸甲基转移酶(tRNA aspartic acid methyltransferase 1, TRDMT1)、YTH N6-甲基腺苷RNA结合蛋白2(YTH N6-methyladenosine RNA binding protein 2, YTHDF2),它们共同用于构建预后模型,其中CDK1、CDKN1A、NOP2、RRM2是预后的危险因素,TCL6、TLR8、TRDMT1、YTHDF2是预后的保护因素,见图2。风险评分=0.117*expCDK1+0.034*expCDKN1A+0.045*expNOP2+0.051*expRRM2−0.069*expTCL6−0.045*expTLR8−0.147*expTRDMT1−0.034*expYTHDF2。
![]() 图 2 预后模型构建及风险评分分布Figure 2 Construction of the prognostic model and distribution of risk scoresA, B: univariate Cox and Lasso regression analyses of prognostic m5C modification-related genes in TCGA cohort; C: distribution of risk scores, survival status, and expression of m5C modification-related genes for each patient in TCGA cohort.
图 2 预后模型构建及风险评分分布Figure 2 Construction of the prognostic model and distribution of risk scoresA, B: univariate Cox and Lasso regression analyses of prognostic m5C modification-related genes in TCGA cohort; C: distribution of risk scores, survival status, and expression of m5C modification-related genes for each patient in TCGA cohort.2.3 预后模型效果评估
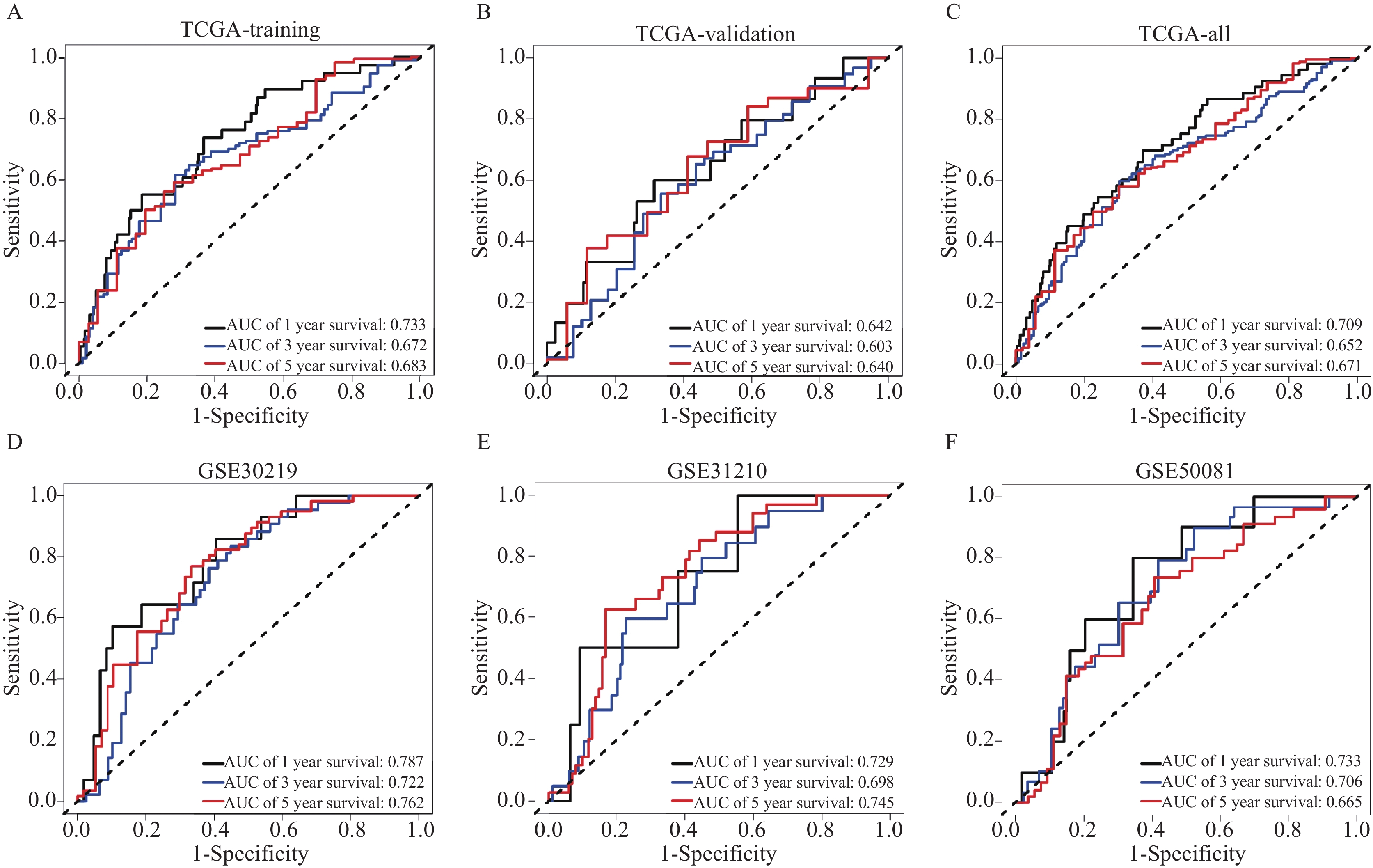
使用Kaplan-Meier生存曲线分析高、低风险组的预后情况。结果显示,高风险组的总体生存率在TCGA训练集、验证集、全集及GSE30219、GSE31210、GSE50081三个测试集中,均比低风险组短,预后较差(均P<0.05),见图3。
![]() 图 3 TCGA训练集、验证集、全集及GEO 3个测试集预后模型的评估(Kaplan-Meier曲线)Figure 3 Evaluation of prognostic model using TCGA training, validation, full, and three GEO test cohorts shown by Kaplan-Meier curveA–F: survival analysis of high- and low-risk patients in TCGA training, validation, all, GSE30219, GSE31210, and GSE50081 cohorts.
图 3 TCGA训练集、验证集、全集及GEO 3个测试集预后模型的评估(Kaplan-Meier曲线)Figure 3 Evaluation of prognostic model using TCGA training, validation, full, and three GEO test cohorts shown by Kaplan-Meier curveA–F: survival analysis of high- and low-risk patients in TCGA training, validation, all, GSE30219, GSE31210, and GSE50081 cohorts.使用ROC曲线评估模型的预测效能,结果提示该模型预测LUAD患者的1年、3年、5年存活率的敏感度和准确性较高,具有较好的预测效能,见图4。
2.4 列线图、校准曲线和决策曲线分析
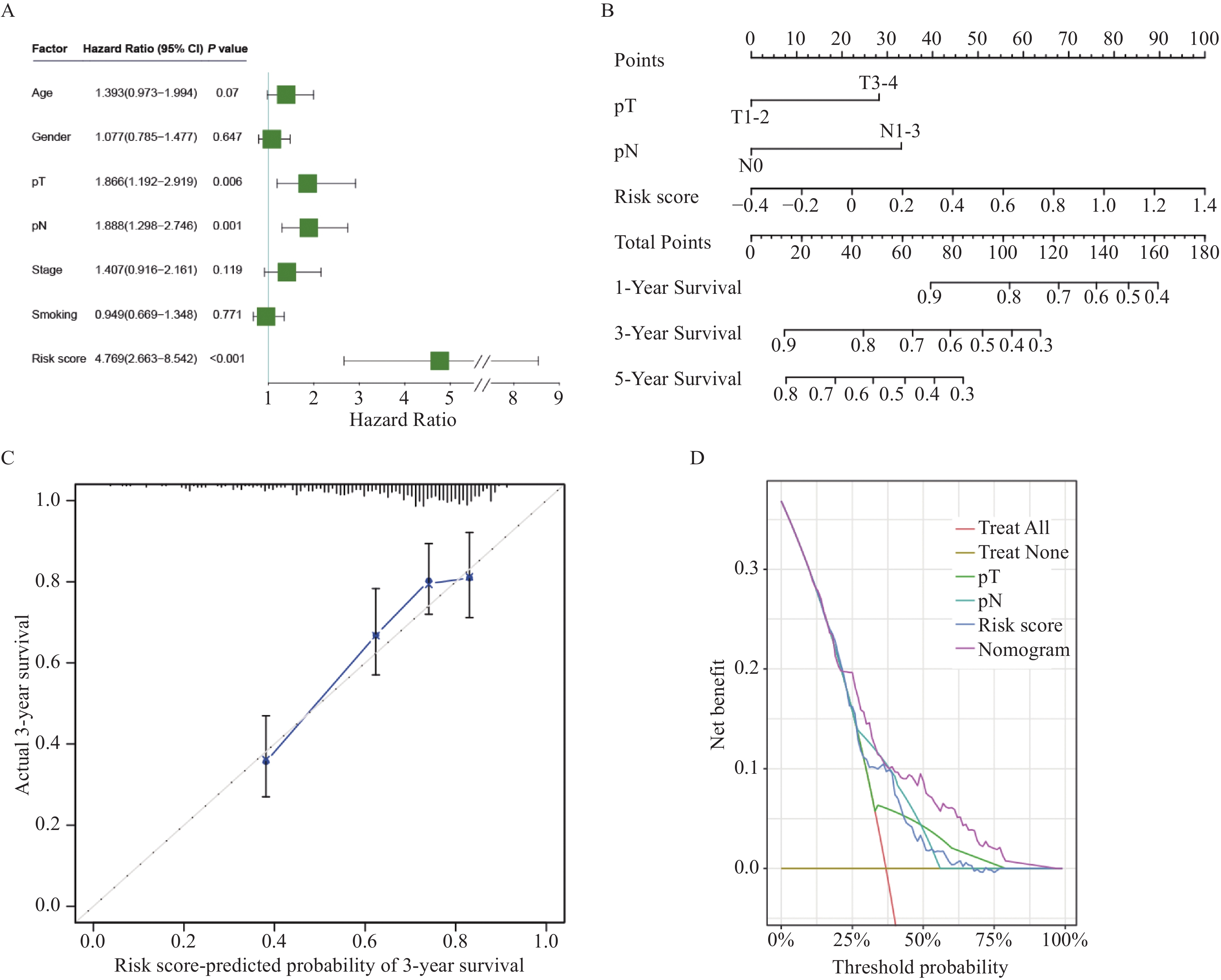
在TCGA数据集中,多因素Cox回归分析结果显示T分期、N分期、风险评分是LUAD患者独立的预后危险因素(均P<0.05),见图5A。纳入上述因素构建列线图模型,见图5B。校准曲线显示,该列线图模型能准确预测患者的3年生存预后情况,见图5C。决策曲线显示,该列线图模型的预测效能,远远高于T分期、N分期及风险评分等因素的预测效能,见图5D。
![]() 图 5 列线图的构建和验证Figure 5 Construction and validation of nomogramA: multivariate Cox analyses of the prognostic risk score and clinical characteristics in the TCGA cohort; B: nomograms for predicting the 1-, 3-, and 5-year survival rates of patients; C: calibration curve to verify the accuracy of the nomogram; D: DCA curves of the OS nomograms of patients.
图 5 列线图的构建和验证Figure 5 Construction and validation of nomogramA: multivariate Cox analyses of the prognostic risk score and clinical characteristics in the TCGA cohort; B: nomograms for predicting the 1-, 3-, and 5-year survival rates of patients; C: calibration curve to verify the accuracy of the nomogram; D: DCA curves of the OS nomograms of patients.2.5 免疫浸润分析
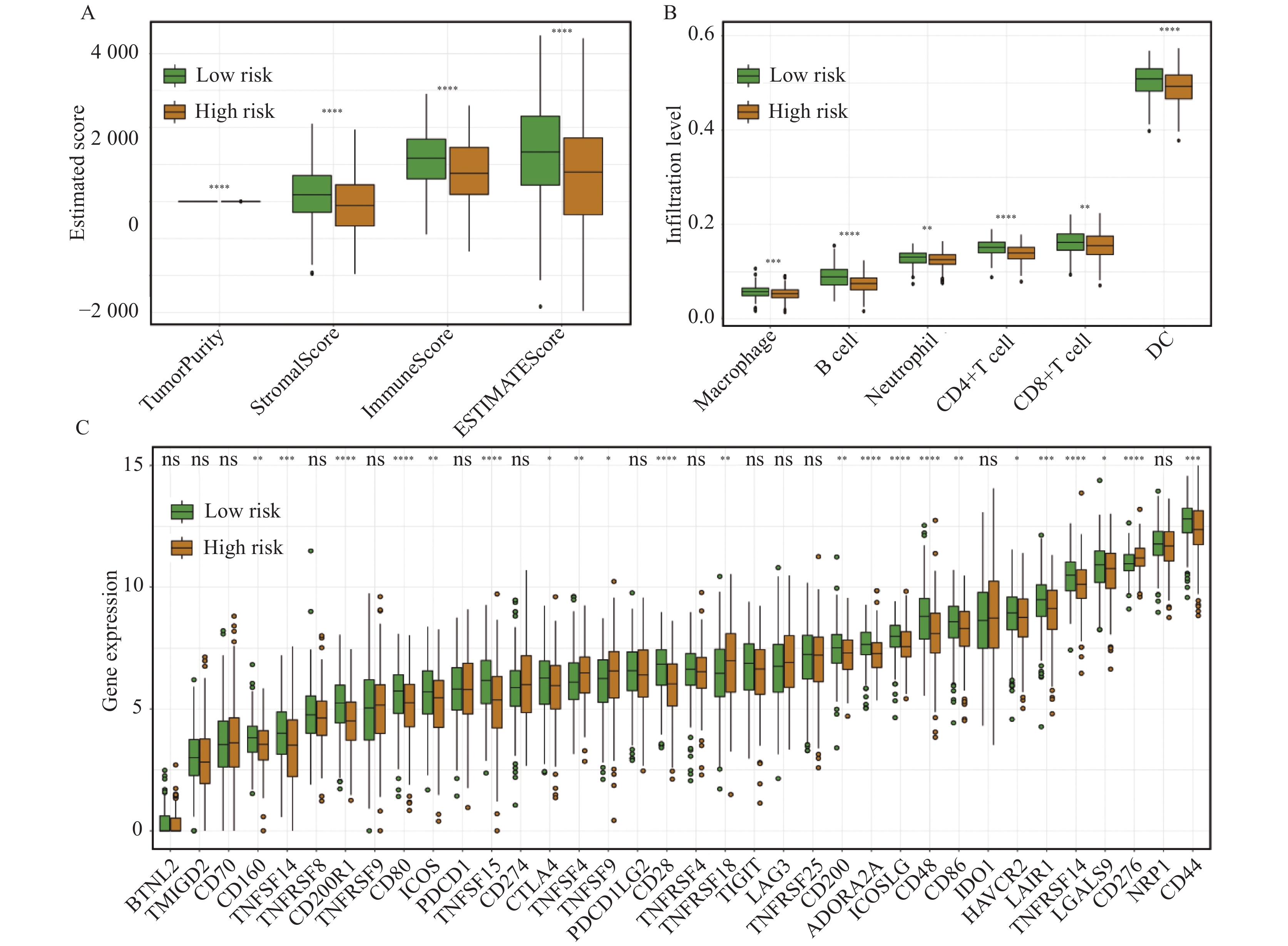
在TCGA数据集中,评估高风险组和低风险组间肿瘤微环境、6种免疫细胞丰度和36种免疫基因表达的差异。高风险组患者的基质评分、免疫评分和ESTIMATE评分明显低于低风险组,说明高风险组患者肿瘤微环境中基质细胞和免疫细胞含量更低,肿瘤细胞含量更高,见图6A。高风险组中巨噬细胞、B细胞、中性粒细胞、CD4+T细胞、CD8+T细胞、树突细胞浸润显著减少,见图6B。两组免疫检查点表达差异结果表明,CD160、TNFSF14、CD200R1、CD80、ICOS、TNFSF15、CTLA4、CD28、CD200、ADORA2A、ICOSLG、CD48、CD86、HAVCR2、LAIR1、TNFRSF14、LGALS9、CD44在低风险组患者中高表达,而TNFSF4、TNFSF9、TNFRSF18、CD276在高风险组患者中高表达,见图6C。
![]() 图 6 高、低风险组免疫浸润分析Figure 6 Comparison of immune infiltration between high- and low-risk groups*: P<0.05, **: P<0.01, ***: P<0.001, ****: P<0.000 1, ns: P>0.05. A, B: ESTIMATE and TIMER analysis showed the differences of immune cell infiltration between high- and low-risk groups; C: difference analysis of immune checkpoints between high- and low-risk groups.
图 6 高、低风险组免疫浸润分析Figure 6 Comparison of immune infiltration between high- and low-risk groups*: P<0.05, **: P<0.01, ***: P<0.001, ****: P<0.000 1, ns: P>0.05. A, B: ESTIMATE and TIMER analysis showed the differences of immune cell infiltration between high- and low-risk groups; C: difference analysis of immune checkpoints between high- and low-risk groups.2.6 风险评分与临床病理特征的关系
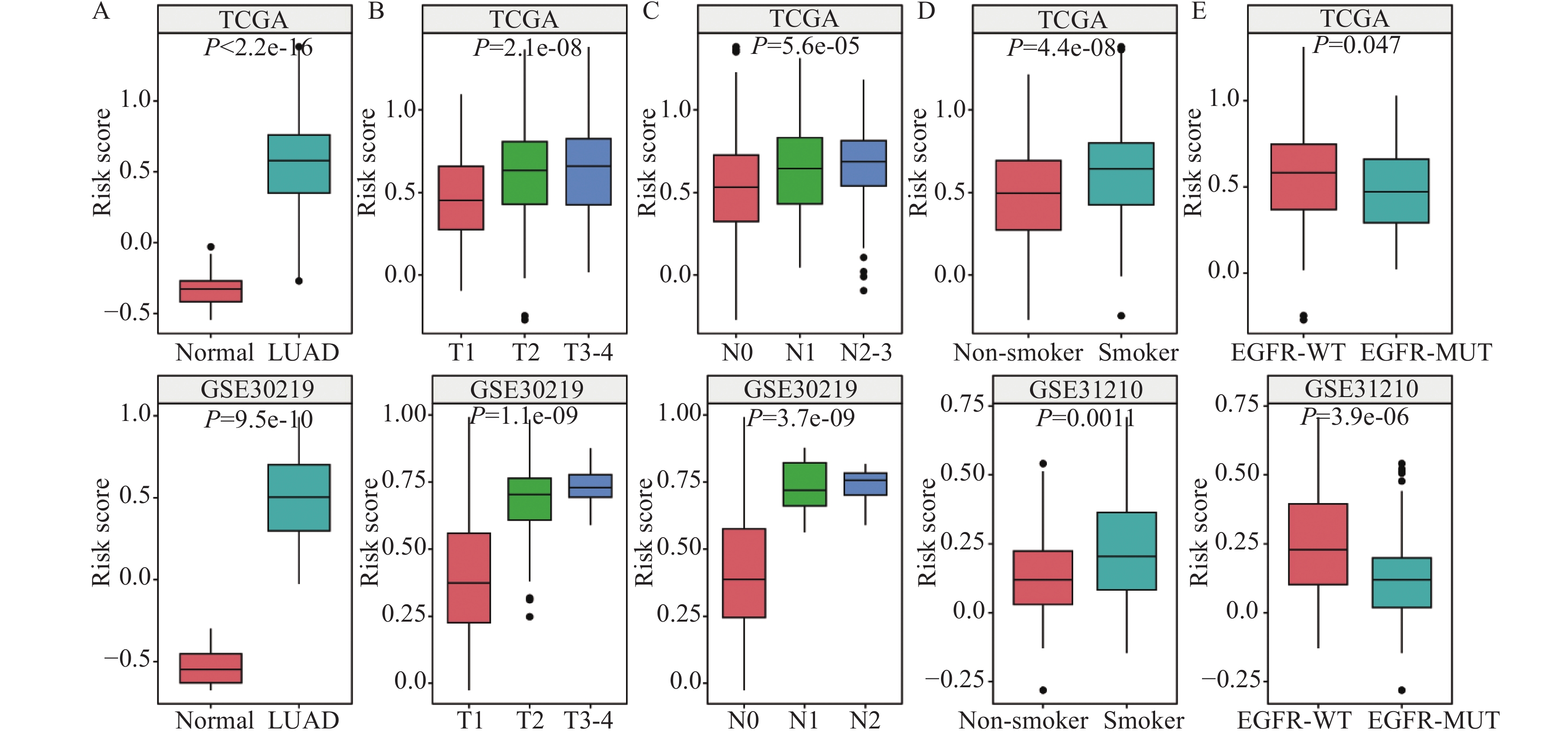
在TCGA和GSE30219数据集中的分析结果表明,LUAD组织中的风险评分均显著高于正常组织,LUAD患者的T分期、N分期越晚风险评分越高,见图7A~C。TCGA和GSE31210数据集的结果表明,吸烟患者风险评分高于不吸烟患者,EGFR野生型患者的风险评分显著高于EGFR突变型患者,见图7D~E。
![]() 图 7 风险评分与患者临床病理特征、吸烟、EGFR基因突变的联系Figure 7 Relationship between risk score and clinicopathological features, smoking, and EGFR mutationA-C: relationship between risk score and clinicopathological features in TCGA and GSE30219 cohorts; D: relationship between risk score and smoking in TCGA and GSE31210 cohorts; E: relationship between risk score and EGFR mutation in TCGA and GSE31210 cohorts.
图 7 风险评分与患者临床病理特征、吸烟、EGFR基因突变的联系Figure 7 Relationship between risk score and clinicopathological features, smoking, and EGFR mutationA-C: relationship between risk score and clinicopathological features in TCGA and GSE30219 cohorts; D: relationship between risk score and smoking in TCGA and GSE31210 cohorts; E: relationship between risk score and EGFR mutation in TCGA and GSE31210 cohorts.3 讨论
LUAD是肺癌的主要亚型之一,具有高发病率和高死亡率的特点。随着检验、影像等诊断技术的不断进步和治疗手段的改善,LUAD患者的整体预后尽管较前有所提升,但其5年生存率仍然较低[11]。LUAD作为一种异质性较强的肿瘤,其发病机制复杂,因此,寻找新的分子标志物和潜在的治疗靶点及建立评估其预后的模型成为当前LUAD研究的重要方向之一。
本研究以m5C修饰相关基因为基础,最终构建了基于CDK1、CDKN1A、NOP2、RRM2、TCL6、TLR8、TRDMT1、YTHDF2等8个基因的预后风险模型,并在3个独立的外部数据集上进行了验证。Cox回归分析显示,风险评分是LUAD患者独立的预后危险因素;纳入肿瘤大小、淋巴结转移及风险评分的列线图模型,可以更准确和更稳定地预测患者的预后状态。
此外,我们还进行了免疫浸润和临床特征相关性分析,以研究该模型对LUAD增殖、转移和免疫微环境的影响。CDK1主要负责G2/M期的转换,与细胞增殖密切相关[12],m5C修饰通过调节CDK1 mRNA的稳定性,促进其在癌细胞中的表达,导致细胞周期加速,增加肿瘤细胞的增殖能力。既往研究表明,CDK1的异常表达与肺癌、乳腺癌、胃癌等多种癌症的恶性进展相关,其高表达往往与不良预后相关[13]。抑制CDK1可能有助于减缓肿瘤的生长,因此CDK1被认为是一个潜在的抗癌靶点。而CDKN1A是细胞周期抑制因子,通常通过抑制CDK1和其他CDKs来阻止细胞从G1期进入S期,在细胞应对DNA损伤时发挥作用[14]。m5C修饰可以调节CDKN1A基因的表达,导致其功能失效或表达降低,从而促使肿瘤细胞逃避细胞周期检查点,加速癌症进展。NOP2属于m5C甲基转移酶家族,能够在rRNA中催化m5C修饰。研究表明NOP2在肝癌中高表达,其可以通过催化c-Myc mRNA的m5C甲基化上调c-Myc的表达,激活肝癌细胞的糖酵解过程,为肿瘤细胞提供生长优势,促进肝癌的发生发展[12]。NOP2敲除可抑制肝癌细胞的增殖和转移,为肝癌的靶向治疗提供潜在新策略。核苷酸还原酶(RR)参与核苷酸代谢,是DNA生物合成、修复和复制所必需的限速酶,哺乳动物的RR包含两种不同的同源二聚体亚基,即大亚基RRM1和小亚基RRM2。RRM2的表达受到m5C修饰的调控,在多种癌症中表现为上调,其过表达不仅促进了肿瘤细胞的DNA合成和增殖能力,还与肿瘤免疫、化疗耐药等密切相关[15]。RRM2在前列腺癌、肺腺癌等癌症中被认为是铁摄取相关代谢基因[16-17],抑制RRM2的活性有望作为一种抗癌治疗策略。TCL6是一种相对较新的癌症相关基因,最早在T细胞白血病患者中被发现,尽管其具体功能尚不完全明确,但有研究发现其可能在细胞周期和细胞分化过程中发挥作用[18]。TLR8是一种重要的模式识别受体,在多种免疫细胞中均有表达,包括单核细胞、巨噬细胞和调节性T细胞等。TLR8能够逆转调节性T细胞免疫抑制功能,具有较强的抗肿瘤作用。TLR8激动剂可导致多种促炎细胞因子的释放,如IL-6、IL-12、TNF-α等,激活先天性免疫[19],m5C修饰可能通过调控TLR8的表达或活性,影响肿瘤细胞的免疫逃逸和微环境变化。根据TLR8作用机制,可与免疫检查点抑制剂和化疗药物等联合使用来提高疗效。既往研究中,TRDMT1被认为是直肠癌患者的保护因子[20]。TRDMT1是m5C修饰主要的甲基转移酶,负责tRNA中的m5C修饰,通过稳定tRNA分子,维持蛋白质合成的正常进行,从而促进肿瘤细胞的快速增殖。YTHDF2是m6A(N6-甲基腺嘌呤)的主要阅读蛋白之一,能够识别并结合m6A修饰的蛋白,与胃癌、肺癌、肝癌等多种肿瘤密切相关,其在不同肿瘤细胞中调控机制不同,亦可能发挥双重作用,既能促进肿瘤细胞增殖和转移,也能抑制肿瘤细胞生长[21]。
本研究加深了我们对LUAD发病机制中RNA甲基化相关调节机制的认识,构建出基于m5C修饰相关基因的LUAD预后预测模型,并通过内部和GEO外部验证集验证了该风险模型的良好预后评估效能。该模型中关键的m5C修饰相关基因在LUAD的发生和发展中发挥了重要的作用,有望成为潜在的治疗靶点。然而本研究仍存在一定的局限性:首先,数据来源于公共数据库,可能存在数据质量和样本代表性的问题;其次,仅针对LUAD进行了分析,未涉及其他的肺癌类型,模型的适用范围有限。未来,我们将收集临床样本进行验证,并通过分子生物学、细胞学等方法进一步探讨m5C修饰相关基因影响肿瘤发生、发展的作用与机制。
Competing interests: The authors declare that they have no competing interests.利益冲突声明:所有作者均声明不存在利益冲突。作者贡献:杨 帆:文章设计、实验设计与撰写王浓燕:文章设计、数据分析方 蒙:数据分析、文章修改章莹姣:文章修改胡海燕:文章设计、文章修改方 鹏:指导及审阅文章 -
-
[1] Sung H, Ferlay J, Siegel R, et al. Global Cancer Statistics 2020: GLOBOCAN Estimates of Incidence and Mortality Worldwide for 36 Cancers in 185 Countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. doi: 10.3322/caac.21660
[2] 国家卫生健康委办公厅. 原发性肺癌诊疗指南(2022年版)[J]. 协和医学杂志, 2022, 13(4): 549-570. [National Health Commission of the People’s Republic of China. Clinical Practice Guideline for Primary Lung Cancer (2022 Version)[J]. Xie He Yi Xue Za Zhi, 2022, 13(4): 549-570.] National Health Commission of the People’s Republic of China. Clinical Practice Guideline for Primary Lung Cancer (2022 Version)[J]. Xie He Yi Xue Za Zhi, 2022, 13(4): 549-570.
[3] Pietro B, Filip S, Angana R, et al. MODOMICS: a database of RNA modification pathways. 2021 update[J]. Nucleic Acids Res, 2021, 50(D1): D231-D235.
[4] Song H, Zhang J, Liu B, et al. Biological roles of RNA m5C modification and its implications in Cancer immunotherapy[J]. Biomark Res, 2022, 10(1): 15. doi: 10.1186/s40364-022-00362-8
[5] Guo G, Wang H, Shi X, et al. Disease Activity-Associated Alteration of mRNA m5C Methylation in CD4+T Cells of Systemic Lupus Erythematosus[J]. Front Cell Dev Biol, 2020, 8: 430. doi: 10.3389/fcell.2020.00430
[6] Chen X, Li A, Sun BF, et al. 5-methylcytosine promotes pathogenesis of bladder cancer through stabilizing mRNAs[J]. Nat Cell Biol, 2019, 21(8): 978-990. doi: 10.1038/s41556-019-0361-y
[7] 陈小梅, 王安奇, 杨积祯, 等. m1A/m5C/m6A/m7G调控基因预测胃癌预后及免疫关联性[J]. 实用医学杂志, 2024, 40(9): 1230-1237. [Chen XM, Wang AQ, Yang JZ, et al. Prognosis and immune correlation analysis of m1A/m5C/m6A/m7G regulated genes in gastric cancer[J]. Shi Yong Yi Xue Za Zhi, 2024, 40(9): 1230-1237.] Chen XM, Wang AQ, Yang JZ, et al. Prognosis and immune correlation analysis of m1A/m5C/m6A/m7G regulated genes in gastric cancer[J]. Shi Yong Yi Xue Za Zhi, 2024, 40(9): 1230-1237.
[8] 胡旭钢, 胡艳, 胡海燕, 等. 基于线粒体代谢相关基因构建和验证肺腺癌预后模型[J]. 浙江医学, 2024, 46(17): 1804-1811, 后插1. [Hu XG, Hu Y, Hu HY, et al. Construction and validation of a prognostic model for predicting lung adenocarcinoma based on mitochondrial metabolism related genes[J]. Zhejiang Yi Xue, 2024, 46(17): 1804-1811, insert 1.] Hu XG, Hu Y, Hu HY, et al. Construction and validation of a prognostic model for predicting lung adenocarcinoma based on mitochondrial metabolism related genes[J]. Zhejiang Yi Xue, 2024, 46(17): 1804-1811, insert 1.
[9] 李梦涵, 肖琼, 高鹏, 等. 基于TCGA数据库的消化道肿瘤LncRNA预后风险评分模型[J]. 肿瘤防治研究, 2022, 49(6): 606-611. [Li MH, Xiao Q, Gao P, et al. LncRNA Prognostic Risk Scoring Model for Gastrointestinal Tumors Based on TCGA Database[J]. Zhong Liu Fang Zhi Yan Jiu, 2022, 49(6): 606-611.] Li MH, Xiao Q, Gao P, et al. LncRNA Prognostic Risk Scoring Model for Gastrointestinal Tumors Based on TCGA Database[J]. Zhong Liu Fang Zhi Yan Jiu, 2022, 49(6): 606-611.
[10] Chen X, Qin Z, Zhu X, et al. Identification and validation of telomerase related lncRNAs signature to predict prognosis and tumor immunotherapy response in bladder cancer[J]. Sci Rep, 2023, 13(1): 21816. doi: 10.1038/s41598-023-49167-1
[11] Xia C, Dong X, Li H, et al. Cancer statistics in China and United States, 2022: profiles, trends, and determinants[J]. Chin Med J (Engl), 2022, 135(5): 584-590. doi: 10.1097/CM9.0000000000002108
[12] Wang Q, Bode AM, Zhang T. Targeting CDK1 in cancer: mechanisms and implications[J]. NPJ Precis Oncol, 2023, 7(1): 58. doi: 10.1038/s41698-023-00407-7
[13] Suski JM, Braun M, Strmiska V, et al. Targeting cell-cycle machinery in cancer[J]. Cancer Cell, 2021, 39(6): 759-778. doi: 10.1016/j.ccell.2021.03.010
[14] Manousakis E, Miralles CM, Esquerda MG, et al. CDKN1A/p21 in Breast Cancer: Part of the Problem, or Part of the Solution?[J]. Int J Mol Sci, 2023, 24(24): 17488. doi: 10.3390/ijms242417488
[15] Zuo Z, Zhou Z, Chang Y, et al. Ribonucleotide reductase M2 (RRM2): Regulation, function and targeting strategy in human cancer[J]. Genes Dis, 2022, 11(1): 218-233.
[16] Ke ZB, You Q, Sun JB, et al. A Novel Ferroptosis-Based Molecular Signature Associated with Biochemical Recurrence-Free Survival and Tumor Immune Microenvironment of Prostate Cancer[J]. Front Cell Dev Biol, 2022, 9: 774625. doi: 10.3389/fcell.2021.774625
[17] Tang B, Xu W, Wang Y, et al. Identification of critical ferroptosis regulators in lung adenocarcinoma that RRM2 facilitates tumor immune infiltration by inhibiting ferroptotic death[J]. Clin Immunol, 2021, 232: 108872. doi: 10.1016/j.clim.2021.108872
[18] Cuadros M, Andrades Á, Coira IF, et al. Expression of the long non-coding RNA TCL6 is associated with clinical outcome in pediatric B-cell acute lymphoblastic leukemia[J]. Blood Cancer J, 2019, 9(12): 93. doi: 10.1038/s41408-019-0258-9
[19] Ohto U, Tanji H, Shimizu T. Structure and function of toll-like receptor 8[J]. Microbes Infect, 2014, 16(4): 273-282. doi: 10.1016/j.micinf.2014.01.007
[20] Zhang R, Gan W, Zong J, et al. Developing an m5C regulator-mediated RNA methylation modification signature to predict prognosis and immunotherapy efficacy in rectal cancer[J]. Front Immunol, 2023, 14: 1054700. doi: 10.3389/fimmu.2023.1054700
[21] 逯涛峰, 杨健, 赵调红, 等. YTHDF2与恶性肿瘤关系的研究进展[J]. 江苏医药, 2022, 48(8): 851-854. [Lu TF, Yang J, Zhao DH, et al. Advances in the study of the relationship between YTHDF2 and malignant tumors[J]. Jiangsu Yi Yao, 2022, 48(8): 851-854.] Lu TF, Yang J, Zhao DH, et al. Advances in the study of the relationship between YTHDF2 and malignant tumors[J]. Jiangsu Yi Yao, 2022, 48(8): 851-854.



 下载:
下载: