Immune Cell-Mediated Effect of Lipid Profile on Colorectal Cancer: A Two-Step, Two-Sample Mendelian Randomization Study
-
摘要:目的
利用两步、双样本孟德尔随机化(MR)方法阐明脂质性状与结直肠癌(CRC)的双向因果关系,同时探讨免疫细胞作为中介因素的介导作用和比例。
方法将研究相关的汇总统计数据进行筛选,主要采用IVW方法对179个脂类与CRC进行双样本、双向MR分析。同时通过贝叶斯加权MR验证因果效应。通过two-step MR分析确定免疫细胞性状是否存在介导作用。通过敏感性分析、异质性分析、水平多效性分析验证因果关系的可靠性。
结果初步确定了9个脂质性状与CRC之间的因果关系,且不存在反向因果效应(P>0.05)。贝叶斯加权MR验证了结果的稳健性(P<0.05)。提示27种免疫细胞与CRC存在因果效应。通过中介分析确定了磷脂酰胆碱(O-18:2_20:4)对CRC的因果作用(OR:0.8579,95%CI=0.7395~0.9952,P=0.0429),CD45RA+ CD4+ T细胞上的CD127的介导比例为9.14%(β=−0.1052,P=0.0155)。
结论脂质性状与CRC存在因果关系,CD45RA+ CD4+ T细胞上的CD127干预有助于磷脂酰胆碱降低CRC的发病风险。
Abstract:ObjectiveTo elucidate the bidirectional causal relationship between lipid profiles and colorectal cancer (CRC) by using the two-sample and two-step Mendelian randomization (MR) methods, and to explore the mediating role and proportion of immune cells as intermediary factors.
MethodsThe pooled statistical data related to the study were screened, and 179 lipids and CRC were analyzed using two-sample and two-step MR with the inverse variance weighted method. Simultaneously, the causal effect was verified via Bayesian weighted MR. Two-step MR analysis was conducted to determine whether a mediated effect was exerted on immune cell traits. Sensitivity, heterogeneity, and pleiotropy analyses were performed to verify the reliability of the study results.
ResultsThe causal relationship between nine lipid traits and CRC was preliminarily identified, and no reverse causal effect was found (P>0.05). The validity of the results was verified via Bayesian weighted MR (P<0.05). Twenty-seven types of immune cells were suggested to exert a causal effect on CRC. The causal effect of phosphatidylcholine (O-18:2_20:4) on CRC was determined via mediation analysis (OR: 0.8579, 95%CI=0.7395-0.9952, P=0.0429). The CD127-mediated proportion on CD45RA+ CD4+ T cells was 9.14% (β=−0.1052, P=0.0155).
ConclusionA causal relationship exists between lipid traits and CRC, and the intervention of CD127 on CD45RA+ CD4+ T cell helps phosphatidylcholine reduce the risk of CRC.
-
Key words:
- Colorectal cancer /
- Mendelian randomization /
- Causality /
- Immune cells /
- Liposome
-
0 引言
结直肠癌(Colorectal cancer, CRC)作为全球第三大最常见恶性肿瘤,2020年死亡率高达90余万人[1]。流行病学研究显示,工业化国家的CRC发病率明显高于较低收入国家[2]。近年来,分子靶向治疗[3-4]和生物免疫治疗[5]也成为CRC治疗的重要手段。这些新兴治疗手段通过针对特定的分子靶点或激活患者自身的免疫系统来攻击癌细胞,具有高效、低毒的优点。即使如此,转移性CRC仍是一种高致死率疾病,5年生存率仅约为14%[6]。因此,理解CRC病理过程、寻找新的治疗靶点或治疗方法尤为重要。
脂质体在细胞生物学中发挥着多重作用,如细胞膜构成、信号转导和激素合成等[7]。观察性研究发现血脂异常作为代谢综合征的重要组成部分,在前列腺癌、直肠癌和肾癌等多种癌症的致癌机制中扮演着举足轻重的角色[8-10]。既往报道显示高甘油三酯血症是男性结肠腺瘤的一个独立危险因素,对其健康构成了潜在的威胁[11]。上述发现虽可能存在混杂因素的干扰,但足以表明脂质体对肿瘤的发生发展机制存在潜在影响。
脂质通过促炎细胞因子调节实现免疫系统的激活,从而广泛应用于癌症治疗[12]。Kheirolomoom等[13]发现脂质体能够产生更高效的免疫细胞激活效应,且特别体现在CD8 T细胞和NK细胞上,进而促进其抗癌作用。同时免疫疗法已被证明是多种癌症的有效治疗方法[14]。
孟德尔随机化(Mendelian randomization,MR)是一种用于研究因果关系的统计方法,广泛应用于遗传流行病学和生物医学研究中。该方法利用随机分配的遗传变异作为工具,来估计某个暴露因素对于特定结果的因果影响[15]。目前已有多项研究通过MR分析方法探讨CRC与不同暴露因素之间的因果关系[16-17]。这为我们深入了解CRC提供了全新的视角。
本研究将单核苷酸多态性(Single nucleotide polymorphism, SNP)视为工具变量(Instrumental variables, IV),通过双样本孟德尔随机化(Two-sample Mendelian randomization, TSMR)方法评估脂质组性状对于CRC结果变量的影响。同时首次纳入免疫细胞特征作为中介因素,利用两步孟德尔随机化(Two-step Mendelian randomization, two-step MR)方法通过系数乘积法评估所介导的效应比例,最后探讨脂质组性状对免疫细胞以及CRC的影响,旨在为预防、诊断和治疗CRC提供新的策略。
1 材料与方法
1.1 研究设计
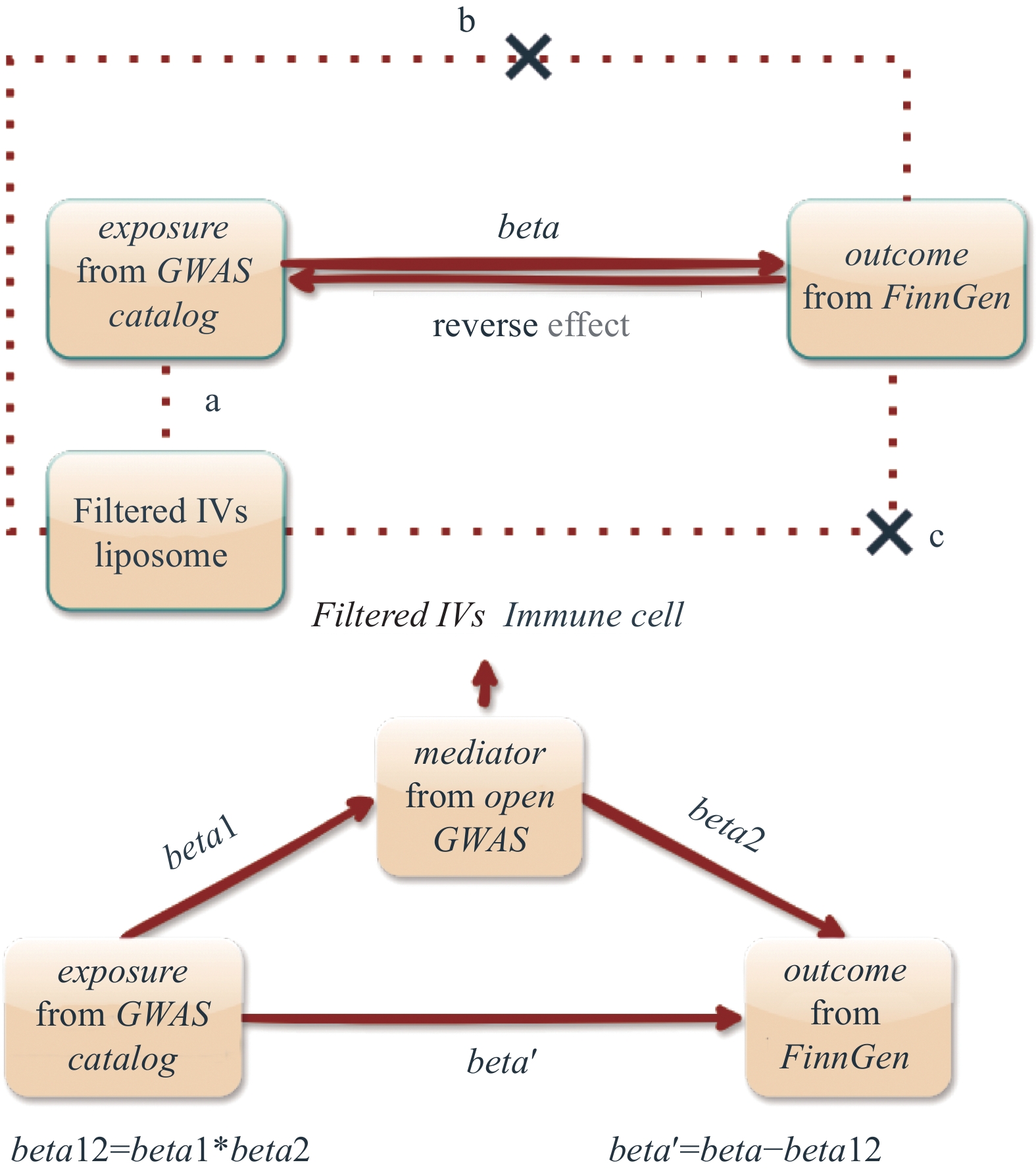
暴露因素对于结局的影响包括总效应和间接效应。前者是指暴露因素直接对结局产生作用,而后者则是通过其他中介因素介导影响结局。因此,我们通过TSMR方法确定脂质体对CRC的直接影响。免疫细胞对CRC的中介作用通过two-step MR确定,见图1。具体来说,我们分别研究了脂质体与免疫细胞、免疫细胞与CRC的因果效应,通过系数乘积法将其确定为影响结局的间接效应。同时,合适的IV需满足以下三个基本假设以减少误差存在的可能性:(1)关联性假设:IV与脂质体密切相关;(2)独立性假设:IV与结果的混杂因素相互独立;(3)排他性假设:IV只能由脂质体通过免疫细胞性状对结局产生作用。
1.2 数据来源
人类血浆脂质组汇总统计数据来源于GWAS catalog数据库(https://www.ebi.ac.uk/)。Ottensmann等[18]对芬兰GeneRISK队列的7 174名个体进行基于质谱的脂质分析,通过鸟枪脂质组学检测到13个脂类共179种脂质,注册ID为GCST90277238至GCST90277416。
英国生物样本库IEU OpenGWAS project数据库(https://gwas.mrcieu.ac.uk/)提供了2020年公开发表的人类全基因组关联研究(Genome-wide association study, GWAS)免疫性状的概括统计数据,注册ID为GCST90001391至GCST90002121[19]。该组免疫细胞队列包括来自欧洲的3 757例个体,具体包括四种特征类型:绝对细胞计数(n=118)、相对细胞计数(n=192)、反映表面抗原水平的中位荧光强度(n=389)和形态参数(n=32)共731种免疫细胞表型。通过撒丁岛队列中结合高分辨率免疫和遗传分析产生SNP数目高达20 143 392个[20]。
CRC的汇总统计数据来自于芬兰FinnGen数据库Consortium R9版本,包括293 646例欧洲个体(病例数为=6 509例,对照数=287 137例)。该数据库包括四十余万芬兰人的遗传信息,旨在利用GWAS和其他遗传数据来深入了解各种疾病的遗传基础。理论上来说,所有GWAS数据均来自不同的联盟或组织,不存在样本重叠。为去除样本重叠偏倚风险,本研究进一步利用连锁不平衡评分回归分析的结果来评估遗传相关性。
1.3 工具变量筛选
在MR分析中,选择SNP作为IV研究暴露因素与结局的因果关系。通过关联性分析挑选全基因组水平上与脂质体、免疫特征、CRC相关的SNP作为IV,筛选阈值条件均为1e-05以满足第一个MR假设。由于基因组位置相近的遗传变异更倾向于共同遗传,致使同时出现在一条染色体的概率高于随机概率。因此使用R包TwoSampleMR中的clump.data()方法限制连锁不平衡条件(千碱基对(kb)=10 000,R2=0.001)以确保SNP的独立性。为消除潜在的重复或回文序列,使用harmonise_data()函数,并设置action=2参数。此外,利用Steiger检验评估反向因果关系的可能性。同时,通过公式$ \mathrm{F}={\mathrm{\beta }}^{2}/{\mathrm{S}\mathrm{E}}^{2} $计算每个SNP的F检验值判断位点与暴露因素之间的相关性强度,若F值<10则被视为弱工具变量筛除[21]。最后,通过检查PhenoScanner (www.phenoscanner.medschl.cam.ac.uk)筛选可能与IV混杂的因素(胆固醇(rs17725246、rs3741298、rs10879574等)、血清代谢物(rs7157785、rs364585、rs58489806等)和吸烟(rs7412、rs10404959、rs4906111等)等并防止这些因素干扰暴露对结果的影响。
1.4 孟德尔随机化分析
利用TSMR方法研究脂质体与CRC之间的潜在关联。具体分析使用R包TwoSampleMR实现。主要采用逆方差加权法(inverse variance weighted, IVW)、MR-Egger回归和加权中位数法(weighted median estimator, WME)等评估因果效应。IVW分析是将每一个SNP方差的倒数作为权重进行拟合,在PFDR<0.05的严格多重假设检验阈值下进行。由于IVW法比其他2种MR方法检验效能更高,主要参考IVW方法作为因果效应评估方法。通过BWMR_updated.R函数实现贝叶斯加权算法,旨在通过比较先验分布与数据驱动的后验分布,验证所揭示的阳性结果是否在贝叶斯加权框架下具备统计显著性。同时,进行反向MR分析消除反向因果干扰。此外通过Bonferroni校正方法避免由多次检验产生的假阳性累积(P<0.05/N),其中N代表进行因果分析的次数。P值在0.05与校正标准区间内仍被视为存在潜在因果关联。最后通过two-step MR评估免疫细胞作为混杂因素的中介效应。为对研究结果更好地进行诠释,本研究将所得的Beta值转换为比值比(odds ratio, OR)并计算95%可信区间(confidence interval, CI)。
基于现有的科学假设和背景知识,同时为避免潜在的偏倚、混杂因素和样本重叠,本研究通过P值、OR值等统计学指标确定了磷脂酰胆碱(phosphatidylcholine, PC)(O-18:2_20:4)-CD45RA+ CD4+ T细胞上的CD127-CRC的two-step MR研究目标。
1.5 质量控制
为确保研究结果的可靠性,进行一系列质量控制措施,包括敏感性分析、异质性检验以及水平基因多效性检验。通过R语言中Leave-one-out()函数将IV逐个剔除后重新进行分析比对结果的影响程度。同时,由于不同的分析平台、实验条件、分析人群等工具变量可能存在因果效应的异质性。通过mr_heterogeneity方法进行Cochran’s Q检验分析,若P>0.05,说明差异无统计学意义,不考虑异质性对于研究结果造成的影响。最后,使用mr_pleiotropy_test方法进行MR多效性残差和和离群值分析通过异常值去除来校正水平多效性的估计值。
2 结果
2.1 工具变量选择
通过预设的筛选标准(P<1e-05、kb=10 000、R2=0.001),在脂质体与CRC的TSMR分析中我们获得了如下IV,SNP数目15~28个不等。为剔除弱工具变量,我们将F统计值的筛选阈值确定为10,见表1。同时,本文提供免疫细胞相关IV的SNP、效应等位基因、等位基因频率以及beta值等详细信息(表略,请扫描OSID码)。
表 1 脂质体与结直肠癌相关的工具变量Table 1 Instrumental variables associated with liposomes and CRCReported trait Data source Sample size Nsnp Fmean Sterol ester (27:1/17:1) levels GWAS catalog 7166 18 22.1621 Ceramide (d40:1) levels GWAS catalog 7117 28 31.1239 Ceramide (d42:1) levels GWAS catalog 7174 27 28.6270 Phosphatidylcholine (18:2_20:1) levels GWAS catalog 5521 17 36.5968 Phosphatidylcholine (O-18:0_16:1) levels GWAS catalog 5959 15 22.9628 Phosphatidylcholine (O-18:2_20:4) levels GWAS catalog 6127 22 25.5984 Phosphatidylethanolamine (O-16:1_22:5) levels GWAS catalog 5609 17 20.8420 Phosphatidylinositol (18:0_18:1) levels GWAS catalog 7162 24 38.1023 Phosphatidylinositol (18:1_18:2) levels GWAS catalog 6489 25 26.3417 2.2 双样本孟德尔随机化分析
2.2.1 脂质体对结直肠癌的因果效应
IVW方法作为主要分析工具,表明三种脂类:磷脂类(n=6)、鞘脂类(n=2)和甾醇脂类(n=1)共9个脂质性状与CRC可能存在因果关系。其中PC(O-18:0_16:1)表现出潜在的正相关性(OR:1.2282,95%CI=1.0189~1.4805,P=0.0311)。该结果在其他分析方法中也得到验证。此外,固醇酯(27:1/17:1)负向调控CRC患病风险(OR:0.8064,95%CI=0.6684~0.9730,P=0.0247),表明它的存在可以降低后者的发生风险,见图2,虽然显著性虽未达到Bonferroni校正水平,但依旧提示潜在的因果关联(P<0.05)。
2.2.2 贝叶斯加权孟德尔随机化
贝叶斯加权MR分析和子集后验聚合增强了研究异质性的解释和计算实用性[22]。鉴于其在因果效应估计中的优势,通过贝叶斯加权MR验证以评估IVW方法计算因果关系的稳健性。结果显示9个脂质体与CRC所得OR值与原始结果方向保持一致,同时P值均小于0.05表示结果具有统计学意义,见图3。
2.2.3 结直肠癌对脂质体的因果效应
将CRC作为暴露因素,脂质性状作为结局进行因果关系分析。结果表明与CRC相关的9个脂质体在反向MR分析中的P值均大于0.05,即CRC的遗传易感性对任何脂质性状均无影响,同时,Steiger检验进一步佐证两者的反向因果关联(表略,请扫描OSID码)。图4显示IVW、MR Egger和WME方法的估值。
2.3 两步孟德尔随机化分析
2.3.1 免疫细胞与结直肠癌的因果效应
通过IVW方法评估731种免疫细胞性状与CRC的因果关系。筛选到淋巴细胞(n=18)、单核细胞(n=4)、树突状细胞(n=2)、粒细胞(n=2)、造血干细胞(n=1)共27种,与CRC相关的免疫细胞特征见图5。结果显示7种免疫细胞对CRC起到保护作用,表明疾病的发生概率与此类免疫性状风险水平负相关,其中CD4+CD8dim T细胞(OR:0.8711,95%CI=0.7732~0.9814,P=0.0233)、CD45RA+ CD4+ T 细胞上的CD127(OR:0.9001,95%CI=0.8266~0.9801,P=0.0155)呈较为明显的负相关关系。同时CRC发生风险与20种免疫细胞呈正向调控关系,其中CD4+CD8+ T细胞百分比T cell最为显著(OR:1.3631,95%CI=1.0782~1.7232,P=0.0096)。
2.3.2 脂质体对免疫细胞的因果效应
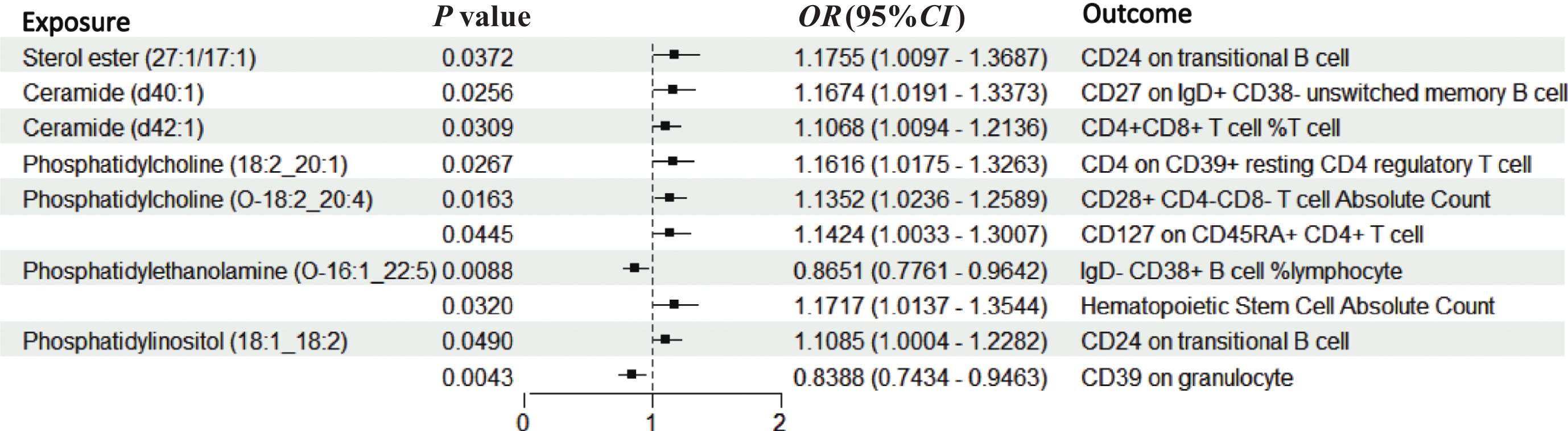
凭借先前研究获得的9个脂质体和27个与CRC相关的免疫细胞性状,再一次进行TSMR分析以确定与脂质体相关的免疫细胞。结果显示共有10个免疫细胞与7个脂质体性状因果相关。其中磷脂酰乙醇胺(O-16:1_22:5)(OR:0.8651,95%CI=0.7761~0.9642,P=0.0088)、磷脂酰肌醇(18:1_18:2)(OR:0.8388,95%CI=0.7434~0.9463,P=0.0043)分别与IgD− CD38+ B cell % lymphocyte、粒细胞上的CD39呈负相关。同时磷脂酰乙醇胺(O-16:1_22:5)(OR:1.1717,95%CI=1.0137~1.3544,P=0.0320)、固醇酯(27:1/17:1)(OR:1.755,95%CI=1.0097~1.3687,P=0.0372)与造血干细胞绝对计数、过渡性B细胞上的CD24均呈显著正相关,见图6。
2.3.3 潜在中介效应计算
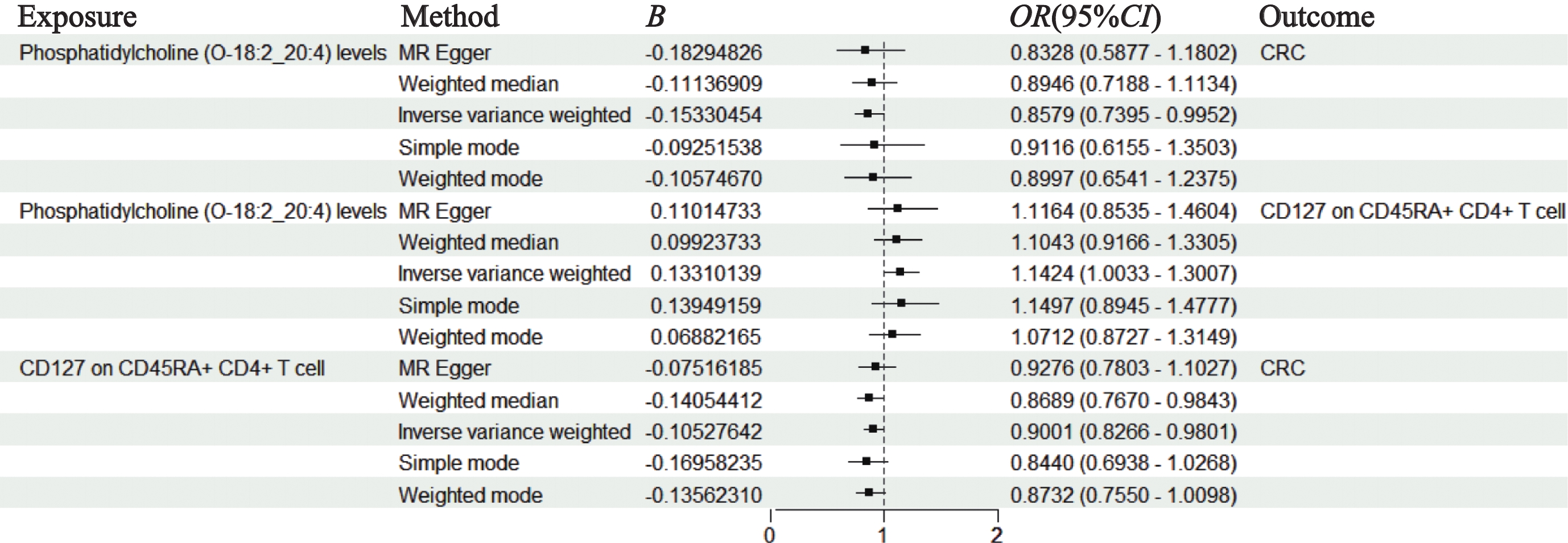
在two-step MR分析中发现研究所选的脂质性状与CRC存在显著的负向调控关系(OR:0.8579,95%CI=0.7395~0.9952,P=0.0429),即PC(O-18:2_20:4)可能对CRC起到关键性保护作用,见图7。同样地,CD45RA+ CD4+ T细胞上的CD127作为本研究所选中介变量同样与结局因素呈负相关(OR:0.9001,95%CI=0.8266~0.9801,P=0.0154)。值得注意的是PC(O-18:2_20:4)与中介变量呈正相关关系,这种正向关系表明随着PC(O-18:2_20:4)水平的增加,CD45RA+ CD4+ T细胞上CD127的表达量也相应增加,可能反映了细胞膜磷脂组成与细胞表面受体表达之间的相互作用,进而影响了T细胞的应答能力和免疫功能。具体来说,PC(O-18:2_20:4)与CD127 on CD45RA+ CD4+ T细胞的效应值为0.1331,后者与CRC的因果分析产生的效应值为−0.1053,由此,通过乘积法获得间接效应值。同时,去除混杂因素的影响,PC(O-18:2_20:4)与CRC的总效应值为−0.1533。结果表明免疫细胞作为中介因素所产生的介导比例为9.14%,见表2。
表 2 CD45RA+ CD4+ T细胞上的CD127在磷脂酰胆碱与CRC因果关系中的介导比例Table 2 Mediating proportion of CD127 on CD45RA+ CD4+ T cells in the causal relationship between phosphatidylcholine and CRCTotal effect βall Indirect effect β1 Indirect effect β2 Mediating effect Intermediate effect ratio P −0.1533 0.1331 −0.1053 −0.014 9.14% 0.0429 2.4 敏感性分析
连锁不平衡评分回归表明,中介分析的样本独立,不存在重叠问题(|遗传相关系数|<0.2)(表略,请扫描OSID码)。通过留一法分析,所纳入研究的IV的效应值和总效应值大小较为接近,未发现对因果关联估计值影响较大的SNP,采用Cochran’s Q检验及其衍生的P值来系统评估估值中的异质性程度,结果显示该检验方法所获P值均大于0.05,即IVW方法估值的核心意义依然得以保持(表略,请扫描OSID码)。为深入探究潜在的多效性水平,我们进一步引入了MR-Egger截距对应的P值作为分析工具,敏感性分析结果均显示P>0.05,即不存在异质性和水平多效性的干扰(表略,请扫描OSID码)。同时,我们还应用了MR-PRESSO全局测试,在分析中识别并剔除了异常值,以校正可能存在的水平多效性。值得一提的是,在所有阳性结果中,剔除这些异常值后的结果与原始分析保持一致(P>0.05),进一步增强了结论的稳健性。
总的来说,通过一系列质量控制的一致性验证,我们加强了初步分析中因果推理的可靠性。
3 讨论
本研究利用丰富的公开遗传数据资源开展了全面的双向TSMR及two-step MR研究。值得一提的是,这是首次探究179种脂质体性状与CRC之间尚未明确的双向因果效应以及潜在的免疫细胞性状中介因素。结果显示5种脂质生物标志物(固醇酯(27:1/17:1)、神经酰胺(d40:1)、神经酰胺(d42:1)、PC(O-18:2_20:4)、磷脂酰乙醇胺(O-16:1_22:5))的水平上升与CRC风险之间存在显著关联,同时,因果关系并非呈现双向特性,即未发现CRC对脂质体相关性状产生显著影响的证据。最后我们分析了9个脂质生物标志物与27个免疫细胞性状的因果关系,表明PC(O-18:2_20:4)与 CD45RA+ CD4+ T上的CD127细胞存在密切关系。Two-step MR结果显示PC(O-18:2_20:4)对降低CRC风险的作用由CD45RA+ CD4+ T细胞上的CD127部分介导(9.14%),这为进一步理解CRC的预防策略提供了新的视角。
脂质组学作为代谢组学的一个重要分支,旨在揭示脂质代谢与机体的生理病理过程之间的关系,为疾病诊断和治疗提供新的视角。多种脂质体通过参与复杂的生物学过程进而影响CRC的形成。LIPID MAPS分类系统将脂质分为八类:脂肪酰基、甘油脂、甘油磷脂、鞘脂、糖脂、聚酮类、甾醇脂质和异戊二烯脂质[23]。Pakiet等[24]通过液相色谱-质谱联用分析发现多种脂质体如神经酰胺、溶血磷脂和甾醇类等显著区分了CRC癌性组织和黏膜组织。Ecker等[25]通过对照试验发现鞘磷脂和三酰基甘油在CRC、CRC非病变组织分布具有差异。同时,PC和磷脂酰乙醇胺作为细胞膜的重要组成成分,其膜含量的变化以及磷脂代谢物水平和脂肪酸谱的改变通常被认为是CRC发生和进展的标志[26]。
PC(O-18:2_20:4),其中“O-18:2_20:4”表示其脂肪酸链的组成。作为一种甘油磷脂,其核心结构由胆碱头基和磷酸基团取代基共同构成[27]。本研究证明PC是CRC因果关系中的关键调控因子,结果并非偶然。Liu等[28]通过建立了Lewis肺癌模型来评估PC的抗癌效果,发现其对原位肿瘤生长和肺转移具有显著的抑制作用。Sakakima团队采用人肝癌细胞系进行体外实验,以评估PC对癌细胞生长的影响,结果同样表明PC对癌细胞具有显著的抑制作用。此外,体内实验也进一步证实含有PC的饮食能够有效减少肝肿瘤结节的数量[29]。同时,Harewood等[30]通过靶向质谱法测量发现包括PC在内的24种脂质性状与较低的CRC风险相关。由于证据仅限于观察性研究,可能受到混杂因素的影响,本研究通过MR分析方法限制混杂因素再次佐证PC对CRC的关键作用。
CD8+T细胞是细胞免疫治疗技术中的关键细胞之一,在肿瘤细胞免疫治疗中发挥着“一线军种”的作用,对肿瘤细胞具有直接的杀伤效果。早在2005年,Pagès等[31]就揭示在没有早期转移迹象的CRC中,Th1细胞以及CD8+T细胞通路的表达显著增强。此后,研究进一步证实,高比例的浸润性CD8+和CD4+T细胞,尤其是Th1细胞,与CRC患者的预后情况存在密切关联[32]。在本研究中,同样证明这些特殊亚群T细胞在CRC中的潜在功效。同时,通过单细胞测序方法,揭示了CRC中T细胞亚群的异质性[33]。Verneau等[34]总结发现树突状细胞介导的肿瘤特异性T细胞对于维持抗肿瘤免疫至关重要,其在肿瘤环境中的存在通常会触发T细胞反应,抑制癌症进展,并有利于患者生存。
Two-step MR因果效应揭示CD45RA+ CD4+ T细胞上的CD127作为中介因素,比例的提升与CRC风险的升高之间存在负相关关系,同时,PC作为暴露因素与该免疫细胞呈显著正向调控关系。CD127,又被称为白细胞介素-7受体α(interleukin-7 receptor alpha,IL-7Rα),对于T细胞的存活、增殖和功能至关重要[35]。Lewis等[36]通过喂养哺乳期大鼠实验证明在脾脏中,磷脂酰胆碱喂养的母细胞表达CD127的比例更高。这与我们的研究结果相吻合,验证了MR分析的可靠性。Wang等[37]通过肿瘤免疫微环境分析揭示IL-7Rα通过调节免疫浸润细胞的比例抑制肺腺癌的发生和进展。IL-7Rα可能成为肿瘤潜在治疗靶点并在癌症受损免疫系统的调节中发挥着关键作用。IL-7Rα的治疗靶向策略涵盖了多个层面,包括受体靶向[38]、NOTCH1靶向[39]、JAK[40]/STAT[41]通路定位以及PI3K/AKT/mTOR[42]通路靶向。Cramer团队[43]发现IL-7Rα信号通路在治疗小儿急性淋巴细胞白血病中扮演着主要的治疗靶点角色。针对该信号通路的治疗靶向策略有望改善一部分患者的预后,为小儿急性淋巴细胞白血病的治疗提供了新方向。即使目前关于CD127与CRC的相关研究尚显不足,凭借现有科学发现依旧能为本次MR分析结果提供有力的正向支持。
本研究通过将免疫细胞视为影响脂质性状与CRC因果关系的中介因素,展开了一项规范完善且统计有效的TSMR,结果显示PC作为暴露因素与CRC的发生发展存在不可忽视的负相关关联。此外,在纳入CD45RA+ CD4+ T细胞上的CD127作为中介因素后提示CRC的发生风险降低,即对该免疫性状的干预可能有助于降低CRC发病率。总之,限制混杂因素后的MR分析结果相对可靠,可为后续CRC病理生理学研究提供依据,推动公共健康事业发展。
本研究具有一定的优势:首先,成功地从多个数据库联盟中下载公开访问大型GWAS统计数据,这些队列涵盖了近30万名欧洲CRC患者以及具有脂质、免疫特征的人群,巨大的样本量提供坚实的数据基础;其次,我们通过双向TSMR、two-step MR分析方法有效地避免了观察性实验存在的混杂因素干扰和反向因果作用;此外,本研究进行了MR分析功效评估,结果显示在合理的参数范围内,具有较高的统计功效,这为发现真实关联提供了稳健的保证;最后,通过贝叶斯加权MR验证因果关系,研究结果相对可靠。但本研究也存在一些局限性:第一,队列数据样本均为欧洲人群,由于种族局限性的存在,可能导致因果关系泛化有限;第二,由于GWAS临床试验数据不包括患者人口学特征信息,本研究未能充分考虑到年龄、性别对关联结果产生的影响;第三,CRC发病年龄通常较晚,因此由其他原因导致较早死亡的人群无法被纳入相关研究。这种由疾病本身及发病时间造成的选择性偏差可能会影响研究对象的代表性,进而导致其与暴露因素的因果关联等估计产生偏差;第四,尽管通过PhenoScanner去除潜在混杂因素,还存在关联强度阈值的设置具有一定主观性,异常严格或宽松都可能导致偏差等问题。最后,在衡量因果效应的同时,也要关注生物学机制。
综上所述,本研究初步探讨了脂质体性状、免疫细胞和CRC之间的因果关系作用机制,发现PC能够通过CD45RA+ CD4+T细胞上的CD127介导降低CRC的发生风险。这将为后续CRC临床实践提供重要指导依据。
Competing interests: The authors declare that they have no competing interests.利益冲突声明:所有作者均声明不存在利益冲突。作者贡献:龚万里:思路设计、数据分析、论文撰写侯雅琪:思路设计、制图、结果验证王钺、李渊、齐荣暄:数据获取、材料收集于琦、章娟:思路设计、资金支持、课题指导及论文审阅修改 -
表 1 脂质体与结直肠癌相关的工具变量
Table 1 Instrumental variables associated with liposomes and CRC
Reported trait Data source Sample size Nsnp Fmean Sterol ester (27:1/17:1) levels GWAS catalog 7166 18 22.1621 Ceramide (d40:1) levels GWAS catalog 7117 28 31.1239 Ceramide (d42:1) levels GWAS catalog 7174 27 28.6270 Phosphatidylcholine (18:2_20:1) levels GWAS catalog 5521 17 36.5968 Phosphatidylcholine (O-18:0_16:1) levels GWAS catalog 5959 15 22.9628 Phosphatidylcholine (O-18:2_20:4) levels GWAS catalog 6127 22 25.5984 Phosphatidylethanolamine (O-16:1_22:5) levels GWAS catalog 5609 17 20.8420 Phosphatidylinositol (18:0_18:1) levels GWAS catalog 7162 24 38.1023 Phosphatidylinositol (18:1_18:2) levels GWAS catalog 6489 25 26.3417 表 2 CD45RA+ CD4+ T细胞上的CD127在磷脂酰胆碱与CRC因果关系中的介导比例
Table 2 Mediating proportion of CD127 on CD45RA+ CD4+ T cells in the causal relationship between phosphatidylcholine and CRC
Total effect βall Indirect effect β1 Indirect effect β2 Mediating effect Intermediate effect ratio P −0.1533 0.1331 −0.1053 −0.014 9.14% 0.0429 -
[1] Sung H, Ferlay J, Siegel RL, et al. Global Cancer Statistics 2020: GLOBOCAN Estimates of Incidence and Mortality Worldwide for 36 Cancers in 185 Countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. doi: 10.3322/caac.21660
[2] Klimeck L, Heisser T, Hoffmeister M, et al. Colorectal cancer: A health and economic problem[J]. Best Pract Res Clin Gastroenterol, 2023, 66: 101839. doi: 10.1016/j.bpg.2023.101839
[3] Wang M, Hu S, Yang J, et al. Arenobufagin inhibits lung metastasis of colorectal cancer by targeting c-MYC/Nrf2 axis[J]. Phytomedicine, 2024, 127: 155391. doi: 10.1016/j.phymed.2024.155391
[4] Zhao H, Ming T, Tang S, et al. Wnt signaling in colorectal cancer: pathogenic role and therapeutic target[J]. Mol Cancer, 2022, 21(1): 144. doi: 10.1186/s12943-022-01616-7
[5] Johdi NA, Sukor NF. Colorectal Cancer Immunotherapy: Options and Strategies[J]. Front Immunol, 2020, 11: 1624. doi: 10.3389/fimmu.2020.01624
[6] Rumpold H, Niedersüß-Beke D, Heiler C, et al. Prediction of mortality in metastatic colorectal cancer in a real-life population: a multicenter explorative analysis[J]. BMC Cancer, 2020, 20(1): 1149. doi: 10.1186/s12885-020-07656-w
[7] Gross RW, Han X. Lipidomics at the interface of structure and function in systems biology[J]. Chem Biol, 2011, 18(3): 284-291. doi: 10.1016/j.chembiol.2011.01.014
[8] Zhang GM, Qin XJ, Zhang HL, et al. Serum lipid profiles: novel biomarkers predicting advanced prostate cancer in patients receiving radical prostatectomy[J]. Asian J Androl, 2015, 17(2): 239-244. doi: 10.4103/1008-682X.142135
[9] Wulaningsih W, Garmo H, Holmberg L, et al. Serum Lipids and the Risk of Gastrointestinal Malignancies in the Swedish AMORIS Study[J]. J Cancer Epidemiol, 2012, 2012: 792034.
[10] Zhang GM, Zhu Y, Luo L, et al. Prevalence of dyslipidaemia in patients with renal cell carcinoma: a case-control study in China[J]. BJU Int, 2014, 113(5b): E75-E81.
[11] Tabuchi M, Kitayama J, Nagawa H. Hypertriglyceridemia is positively correlated with the development of colorectal tubular adenoma in Japanese men[J]. World J Gastroenterol, 2006, 12(8): 1261-1264. doi: 10.3748/wjg.v12.i8.1261
[12] Yamamoto S, Ishida T, Inoue A, et al. HEPC-based liposomes trigger cytokine release from peripheral blood cells: effects of liposomal size, dose and lipid composition[J]. Int J Pharm, 2002, 236(1-2): 125-133. doi: 10.1016/S0378-5173(02)00026-1
[13] Kheirolomoom A, Ingham ES, Mahakian LM, et al. CpG expedites regression of local and systemic tumors when combined with activatable nanodelivery[J]. J Control Release, 2015, 220(Pt A): 253-264.
[14] Chen DS, Mellman I. Elements of cancer immunity and the cancer-immune set point[J]. Nature, 2017, 541(7637): 321-330. doi: 10.1038/nature21349
[15] Sekula P, Del Greco MF, Pattaro C, et al. Mendelian Randomization as an Approach to Assess Causality Using Observational Data[J]. J Am Soc Nephrol, 2016, 27(11): 3253-3265. doi: 10.1681/ASN.2016010098
[16] Zhan ZQ, Huang ZM, Zeng RQ, et al. Association between Atopic Dermatitis and Colorectal Cancer: TET2 as a Shared Gene Signature and Prognostic Biomarker[J]. J Cancer, 2024, 15(5): 1414-1428. doi: 10.7150/jca.92238
[17] Zhang C, Huang W, Niu W, et al. Five genes identified as prognostic markers for colorectal cancer through the integration of genome-wide association study and expression quantitative trait loci data[J]. Per Med, 2024, 21(2): 103-116. doi: 10.2217/pme-2023-0103
[18] Ottensmann L, Tabassum R, Ruotsalainen SE, et al. Genome-wide association analysis of plasma lipidome identifies 495 genetic associations[J]. Nat Commun, 2023, 14(1): 6934. doi: 10.1038/s41467-023-42532-8
[19] Orrù V, Steri M, Sidore C, et al. Complex genetic signatures in immune cells underlie autoimmunity and inform therapy[J]. Nat Genet, 2020, 52(10): 1036-1045. doi: 10.1038/s41588-020-0684-4
[20] Sidore C, Busonero F, Maschio A, et al. Genome sequencing elucidates Sardinian genetic architecture and augments association analyses for lipid and blood inflammatory markers[J]. Nat Genet, 2015, 47(11): 1272-1281. doi: 10.1038/ng.3368
[21] Bowden J, Del Greco M F, Minelli C, et al. Assessing the suitability of summary data for two-sample Mendelian randomization analyses using MR-Egger regression: the role of the I2 statistic[J]. Int J Epidemiol, 2016, 45(6): 1961-1974.
[22] Zou L, Guo H, Berzuini C. Bayesian mendelian randomization with study heterogeneity and data partitioning for large studies[J]. BMC Med Res Methodol, 2022, 22(1): 162. doi: 10.1186/s12874-022-01619-4
[23] Fahy E, Cotter D, Sud M, et al. Lipid classification, structures and tools[J]. Biochim Biophys Acta, 2011, 1811(11): 637-647. doi: 10.1016/j.bbalip.2011.06.009
[24] Pakiet A, Sikora K, Kobiela J, et al. Alterations in complex lipids in tumor tissue of patients with colorectal cancer[J]. Lipids Health Dis, 2021, 20(1): 85. doi: 10.1186/s12944-021-01512-x
[25] Ecker J, Benedetti E, Kindt ASD, et al. The Colorectal Cancer Lipidome: Identification of a Robust Tumor-Specific Lipid Species Signature[J]. Gastroenterology, 2021, 161(3): 910-923. e19.
[26] Stoica C, Ferreira AK, Hannan K, et al. Bilayer Forming Phospholipids as Targets for Cancer Therapy[J]. Int J Mol Sci, 2022, 23(9): 5266. doi: 10.3390/ijms23095266
[27] Leibe R, Fritsch-Decker S, Gussmann F, et al. Key Role of Choline Head Groups in Large Unilamellar Phospholipid Vesicles for the Interaction with and Rupture by Silica Nanoparticles[J]. Small, 2023, 19(34): e2207593. doi: 10.1002/smll.202207593
[28] Liu Y, Tian Y, Cai W, et al. DHA/EPA-Enriched Phosphatidylcholine Suppresses Tumor Growth and Metastasis via Activating Peroxisome Proliferator-Activated Receptor gamma in Lewis Lung Cancer Mice[J]. J Agric Food Chem, 2021, 69(2): 676-685. doi: 10.1021/acs.jafc.0c06890
[29] Sakakima Y, Hayakawa A, Nagasaka T, et al. Prevention of hepatocarcinogenesis with phosphatidylcholine and menaquinone-4: in vitro and in vivo experiments[J]. J Hepatol, 2007, 47(1): 83-92. doi: 10.1016/j.jhep.2007.01.030
[30] Harewood R, Rothwell JA, Bešević J, et al. Association between pre-diagnostic circulating lipid metabolites and colorectal cancer risk: a nested case-control study in the European Prospective Investigation into Cancer and Nutrition (EPIC)[J]. EBioMedicine, 2024, 101: 105024. doi: 10.1016/j.ebiom.2024.105024
[31] Pagès F, Berger A, Camus M, et al. Effector memory T cells, early metastasis, and survival in colorectal cancer[J]. N Engl J Med, 2005, 353(25): 2654-2666. doi: 10.1056/NEJMoa051424
[32] Lhuillier C, Rudqvist NP, Yamazaki T, et al. Radiotherapy-exposed CD8+ and CD4+ neoantigens enhance tumor control[J]. J Clin Invest, 2021, 131(5): e138740. doi: 10.1172/JCI138740
[33] Mei Y, Xiao W, Hu H, et al. Single-cell analyses reveal suppressive tumor microenvironment of human colorectal cancer[J]. Clin Transl Med, 2021, 11(6): e422. doi: 10.1002/ctm2.422
[34] Verneau J, Sautés-Fridman C, Sun CM. Dendritic cells in the tumor microenvironment: prognostic and theranostic impact[J]. Semin Immunol, 2020, 48: 101410. doi: 10.1016/j.smim.2020.101410
[35] Leung GA, Cool T, Valencia CH, et al. The lymphoid-associated interleukin 7 receptor (IL7R) regulates tissue-resident macrophage development[J]. Development, 2019, 146(14): dev176180. doi: 10.1242/dev.176180
[36] Lewis ED, Richard C, Goruk S, et al. Feeding a Mixture of Choline Forms during Lactation Improves Offspring Growth and Maternal Lymphocyte Response to Ex Vivo Immune Challenges[J]. Nutrients, 2017, 9(7): 713. doi: 10.3390/nu9070713
[37] Wang X, Chang S, Wang T, et al. IL7R Is Correlated With Immune Cell Infiltration in the Tumor Microenvironment of Lung Adenocarcinoma[J]. Front Pharmacol, 2022, 13: 857289. doi: 10.3389/fphar.2022.857289
[38] Mansour MR, Reed C, Eisenberg AR, et al. Targeting oncogenic interleukin-7 receptor signalling with N-acetylcysteine in T cell acute lymphoblastic leukaemia[J]. Br J Haematol, 2015, 168(2): 230-238. doi: 10.1111/bjh.13115
[39] Tosello V, Ferrando AA. The NOTCH signaling pathway: role in the pathogenesis of T-cell acute lymphoblastic leukemia and implication for therapy[J]. Ther Adv Hematol, 2013, 4(3): 199-210. doi: 10.1177/2040620712471368
[40] Santos FP, Verstovsek S. JAK2 inhibitors: what's the true therapeutic potential?[J] Blood Rev, 2011, 25(2): 53-63.
[41] Dorritie KA, McCubrey JA, Johnson DE. STAT transcription factors in hematopoiesis and leukemogenesis: opportunities for therapeutic intervention[J]. Leukemia, 2014, 28(2): 248-257. doi: 10.1038/leu.2013.192
[42] Rodon J, Dienstmann R, Serra V, et al. Development of PI3K inhibitors: lessons learned from early clinical trials[J]. Nat Rev Clin Oncol, 2013, 10(3): 143-153. doi: 10.1038/nrclinonc.2013.10
[43] Cramer SD, Aplan PD, Durum SK. Therapeutic targeting of IL-7Ralpha signaling pathways in ALL treatment[J]. Blood, 2016, 128(4): 473-478. doi: 10.1182/blood-2016-03-679209



 下载:
下载: