Influence of LncRNA NKILA on Biological Behavior of Malignant Tumors and Related Mechanism
-
摘要:
NKILA(NF-kappaB interacting lncRNA)是一种新近发现的长链非编码RNA,其在诸多恶性肿瘤中呈现异常表达。现有研究已证实NKILA主要通过NF-κB信号通路参与调控肿瘤的发生和发展,对肿瘤的诊断、治疗和患者预后的评估都有重要意义。本文将对NKILA在多种肿瘤中的异常表达情况和生物学功能作一综述,并对不同肿瘤中NKILA调控恶性生物学行为的上下游机制进行总结与归纳。
Abstract:NKILA is a kind of newly-discovered lncRNA whose expression is aberrant in diverse malignant tumors. The existing researches have confirmed that NKILA participates in the occurrence and development of tumors mainly by regulating the NF-κB signaling pathway, and has significance to the cancer diagnosis, treatment and prognostic evaluation of patients. This article reviews the abnormal expressions and biological effects of NKILA, and the up- and down-stream mechanisms of NKILA regulating malignant biological behavior in different cancers.
-
Key words:
- Long non-coding RNA /
- NKILA /
- Tumors /
- Malignant biological behavior
-
0 引言
据GLOBOCAN 2020数据报道,乳腺癌(breast cancer, BC)是世界上最常见的恶性肿瘤[1]。随着经济的发展,乳腺癌的发病率和死亡率急剧上升[2]。2020年,中国女性乳腺癌新发病例数约占全球的18.41%,其中多达40%的患者发现时已进展为浸润性乳腺癌[3]。近年来,以雌激素信号通路为靶点的多种药物被用于治疗雌激素受体(ER)阳性的乳腺癌,如选择性雌激素受体调节剂(SERM)(如他莫西芬)、雌激素受体拮抗剂(如氟维斯通)、芳香化酶抑制剂(AI),包括可逆非甾体药物(如来曲唑、阿那曲唑)和不可逆非甾体药物(如依西美坦)[4]。
G蛋白偶联雌激素受体(GPER),原名G蛋白偶联受体30(GPR30),是一种新发现的选择性雌激素受体。与传统的核受体ERα和ERβ不同,GPER是一种七跨膜结构域蛋白,主要介导快速的非基因组反应[5-8]。研究发现,传统的雌激素受体调节剂或拮抗剂,如他莫昔芬和氟维司琼是GPER的兴奋剂[9]。许多研究表明,GPER的异常表达与乳腺癌、子宫内膜癌和卵巢癌的发生和发展密切相关[10-12]。
Filardo等通过免疫组织化学分析了GPER表达与乳腺癌症受体类型和临床病理参数的关系,结果GPER高表达水平与肿瘤大小、远处转移和HER2过度表达正相关,表明GPER高表达可能对癌症有独立影响[13]。随后研究发现GPER表达水平与术后仅接受他莫昔芬治疗的患者无复发生存率呈负相关[14]。Yu等报道GPER表达水平与更高的磷酸化-ERK1/2水平、分期和更差的临床结果呈正相关[15]。然而,Chen等报道GPER与更高等级、分期或淋巴结转移呈负相关[16]。可见,GPER表达水平高常与乳腺癌的临床病理学特征相关,如肿瘤大小、淋巴结转移、远处转移、组织学分级和激素受体状态等,但结论仍存在争议。因此,目前有必要结合现有数据来系统评估GPER表达在乳腺癌患者中的预后价值。
1 资料与方法
1.1 搜索策略和研究选择
评估GPER表达与乳腺癌患者生存结局或临床病理特征相关性的相关文章摘自PubMed、EMBASE、Science和中国国家知识基础设施(CNKI)数据库,更新至2022年11月24日。搜索策略使用了以下组合术语:“GPER” or “CMKRL2” or “FEG-1” or “G-protein coupled estrogen receptor”, “breast cancer” or “breast neoplasm” or “breast tumor”。此外,还对合格研究的参考列表进行了进一步的手动搜索,以确定其他相关研究。最初没有采用较低的日期或语言限制,但对于全文审查和数据分析,最终只包括英文或中文的论文。为了尽量减少搜索过程中产生的偏差,扫描了所有相关文章中的参考文献,以确定其他可能适用的报告。
本文收集了通过免疫组织化学(IHC)检测手术切除的原发性BC患者肿瘤组织(癌组织样而非血样)中GPER蛋白表达(非mRNA表达)对预后影响的已发表研究的完整和全面数据。筛选GPER表达与生存结局或临床病理特征之间的相关性研究。对于GPER表达和生存结果,必须满足额外的标准以确保本文的高质量:(1)完整的队列研究论文,评估了GPER蛋白表达与BC临床结果,包括总生存率(OS)、无病生存率(DFS)、无复发生存率(RFS)、无远处疾病生存率(DDFS)之间的关系;(2)风险比(HR)和95%3可从论文中获得,或使用Parmar, Williamson和Tierney基于论文信息开发的方法进行计算;(3)文章的语言为英文和中文。对于GPER表达和临床病理特征,纳入标准:(1)病例对照研究论文,评估GPER蛋白表达与BC临床病理特征的关系;(2)文中的数据可用于计算OR和95%CI。排除标准:(1)动物模型的评论、信件或实验;(2)缺乏HR或OR的关键信息;(3)研究的是GPER多态性而不是表达;(4)文章的语言非英文和中文。当一位作者报告了两份或更多关于潜在同一患者群体时,仅包括最近或完整的研究。合格文献的确认第一步是筛选标题和摘要,第二步是对整篇文章进行回顾。两名评审员独立确定研究资格,通过协商一致解决分歧。
1.2 荟萃分析中使用的生存结果和临床病理特征
OS和与乳腺癌复发相对风险相关的数据被认为是我们对BC患者GPER表达与预后之间关系荟萃分析的临床结果。许多研究评估了OS和DFS结果,或RFS/DDFS。每个结果定义如下:OS,从诊断到死亡或随访结束的时间;DFS,从诊断之日到BC首次远处转移或局部复发或随访结束的时间;RFS,从诊断之日到复发性BC或随访结束;DDFS,从诊断之日到首次远处转移或随访结束。临床病理特征涉及TNM分期、肿瘤大小、淋巴结状态、远处转移、组织学分级、雌激素受体状态和孕激素受体状态。根据BC的TNM分期分类确定TNM分期、肿瘤大小、淋巴结状态和远处转移。根据Scaff-Bloom-Richardson组织学分级系统评估组织学分级。
1.3 数据提取
从文章中提取的数据包括:第一作者姓名、发表年份、国家、患者的中位年龄、中位随访时间、研究样本量、GPER阳性百分比、生存结局、HR估计方法、生存分析方法、HR和95%CI、涉及的临床病理特征、OR,如果原始研究中未提及上述信息,则该项目被视为“未报告(NR)”。通过辩论和协商解决研究过程中的不一致。
1.4 质量评估
根据Cochrane非随机研究方法工作组推荐的纽卡斯尔-渥太华质量评估量表(NOS)独立评估研究质量,两名研究者独立评估两份研究。
1.5 统计学方法
所有统计计算均使用Review Manager 5.4.1。HRs和95%CI用作有效值,以测量GPER表达对BC患者生存的影响。一些纳入的研究直接提供了HR和95%CI,当这些统计变量没有明确给出时,我们使用Parmar、Williamson和Tierney。可用数据包括事件总数、每组患者数、对数秩统计量及其P值或O-E统计量(观察事件数与预期事件数之差)。如果唯一现存的生存数据是图形形式的,Kaplan-Meier生存曲线由两名独立研究人员通过Engauge Digitizer version 4.1(free software downloaded from http://sourceforge.net)来进行分析计算。通过向作者发送电子邮件获得荟萃分析所需的额外信息和数据,包括没有文章或Kaplan-Meier生存疗法的数据。此外,当单变量和多变量分析都可用时,选择后者进行汇总,因为BC的生存结果受到多种因素的影响。
使用OR及其95%CI组合评估GPER表达与临床病理特征之间的相关性。关键暴露变量为阳性或阴性GPER表达,对照组为阴性。相关的结果变量定义为是否存在高TNM分期、大体积肿瘤、淋巴结浸润、远处转移、高组织学分级、雌激素受体阳性表达和孕激素受体阳性表达。HR或OR > 1并且其相应的95%CI和1之间没有重叠意味着GPER阳性表达组的生存率较低,或者GPER表达与临床病理特征之间存在显著关联。
研究之间的异质性通过基于卡方的Q统计检验进行评估。Q检验的P > 0.10表明研究之间缺乏异质性。同时统计I2=0~40%,无或中度异质性;I2 > 40%,显著异质性,以量化研究异质性导致的总变异比例。如果没有显著的异质性,则使用固定效应模型。否则,使用随机效应模型。通过漏斗图的目视检查评估潜在的发表偏倚,其中P < 0.05为差异有统计学意义。通过连续省略的个体研究进行敏感性分析,以检查汇总结果的稳定性。
2 结果
2.1 研究特点
研究共检索了11 995篇文章。通过阅读标题和摘要,排除了非人类的研究、不适当的文章类型和不相关的文章,从而得出69篇文章。在排除不相关的研究、重叠的研究和没有计算HR或OR信息的研究后,9篇文章最终符合GPER表达和生存结果的纳入标准[14, 17-24]。共有13篇文章涉及GPER表达和临床病理特征[13-14, 17-23, 25-28]。研究选择过程见图 1。
乳腺癌生存结果中包括的保留研究主要特征总结见表 1。3项研究评估了来自中国、德国(2例)、瑞士(1例)、瑞典(1例)、斯德哥尔摩(1例)和英国(1例)的患者。6项研究用OS来评估GPER表达在乳腺癌症患者中的预后价值,五项研究用DFS/RFS/DDFS作为指标。共纳入4 500例患者,每项研究的患者数量从161例到1 244例不等。在8项研究中,HR和95%CI直接从原始文章中获得。数据是根据一项单独研究中的可用信息计算得出的。两篇文章获得9个NOS量表,四篇文章获得8分,两篇文章得到7分,一篇文章获得6分。
表 1 荟萃分析中合格生存结果的研究特征Table 1 Characteristics of eligible studies for survival outcomes in meta-analysis
乳腺癌患者临床病理特征中包括的保留研究的主要特征见表 2。4项研究评估了来自中国、德国(3例)、美国(2例)、瑞士(1例)、瑞典(1例)、斯德哥尔摩(1例)和英国(1例)的患者。共纳入5 798名患者,每项研究的患者数量从74例到1 244例不等。GPER的阳性表达率为19.3%~79.6%。13篇文章包括GPER表达对肿瘤大小的影响,11篇文章涉及淋巴结状态,3篇文章涉及远处转移,2篇文章包括TNM分期,14篇文章包括组织学分级信息,10篇文章包括雌激素受体状态,10篇文章包含孕激素受体状态信息。2项研究获得9份NOS量表,5项研究获得8份,5项试验获得7份,1项研究获得6份。
表 2 荟萃分析中临床病理特征的合格研究特征Table 2 Characteristics of eligible studies for clinicopathological features in meta-analysis
2.2 GPER表达对BC患者OS和DFS/RFS的影响
使用随机效应模型的森林图和HR汇总结果见图 2。对评估GPER表达与OS相关性的6项研究进行荟萃分析。合并HR为0.77(95%3: 0.49~1.22, Z=1.10, P=0.27),存在异质性(I2=79%, P=0.0002),见图 2A。5项研究评估了GPER表达与DFS/RFS/DDFS的相关性。合并HR为1.03(95%CI: 0.64~1.65, Z=0.13, P=0.90),存在异质性(I2=75%, P=0.003),见图 2B。这些结果表明GPER表达与BC患者的预后无显著相关性,GPER表达不是BC患者的预后因素。
2.3 GPER表达对BC患者临床病理特征的影响
GPER过度表达与BC临床病理特征的相关性见表 3。2项研究评估了GPER表达与TNM分期之间的关系,见图 3,合并HR为0.31(95%CI: 0.06~0.55, Z=2.43, P=0.02),无异质性(I2=0, P=0.44)。3项研究评估了GPER表达与远处转移的相关性,见图 4,合并HR为6.82(95%CI: 1.89~24.55, Z=2.94, P=0.003),存在异质性(I2=91%, P < 0.00001)。13项研究评估了GPER表达与组织学分级的相关性,见图 5,合并HR为-0.09(95%CI: -0.16~0.01, Z=2.16, P=0.03),存在异质性(I2=76%, P < 0.0001)。10项研究评估了GPER表达和ER表达之间的关联,见图 6,合并HR为1.77(95%CI: 1.15~2.72, Z=2.59, P=0.009),存在异质性(I2=80%, P < 0.0001)。10项研究评估了GPER表达和PR表达之间的关联,见图 7,合并HR为1.36(95%CI: 1.00~1.84, Z=1.95, P=0.005),存在异质性(I2=70%, P=0.0004)。GPER表达与肿瘤大小或淋巴结转移无显著相关性。
表 3 GPER过度表达与癌症临床病理特征相关性的Meta分析Table 3 Meta-analysis of the association between GPER overexpression and clinicopathological features of breast cancer patients
3 讨论
近年来,乳腺癌在治疗方面取得了重大进展,但其预后仍然不佳。许多研究表明,GPER在包括BC的各种人类癌症组织中过度表达。关于GPER在乳腺癌中的预后意义,许多报道结果不一致。本荟萃分析的结果表明,GPER对乳腺癌的预后影响无统计学意义,GPER对于乳腺癌来说可能不是独立的预后因素。此外,GPER表达与TNM分期、远处转移、组织学分级、雌激素受体状态和孕激素受体状态显著相关。根据我们的结果,需要通过多变量分析进行更大规模的前瞻性研究,并考虑众所周知的BC预后因素。
GPER作为一种7-跨膜G蛋白偶联受体,主要存在于细胞的内膜中,包括内质网、高尔基体和质膜[29]。GPER参与雌激素介导的多种生理和病理作用,如调节细胞增殖、凋亡、迁移、免疫应答和代谢等[30]。部分研究发现,GPER上调原癌基因c-Fos的表达,促进乳腺癌细胞的生长[31];GPER还参与上调基因Snail的表达,诱导乳腺癌上皮-间充质转变等[32]。临床和试验数据表明,GPER信号转导在乳腺癌中起着重要作用,但GPER在乳腺癌的预后作用目前仍存在争议。可能的原因包括以下几点:首先,GPER的作用在肿瘤发展的不同阶段差异很大;其次,GPER的生物学效应与癌细胞的氧气水平密切相关;第三,GPER在不同乳腺癌分子亚型可能表现出不同的机制;第四,雌激素生理作用的复杂性和各种类固醇受体之间潜在的串扰可能会影响对GPER预后价值的判断。因此,区分个体的分子和表型亚组对于确定GPER在乳腺癌中的贡献可能至关重要。大量GPER在乳腺癌中作用的相关信息也表明,GPER有望作为乳腺癌的有效预测因子和治疗靶点。
我们对纳入研究进行了严格筛选,以避免一些选择偏差,确保研究的质量和可比性。我们还通过尽可能完整地使用PubMed、Ovid、EMBASE、Web of Science和CNKI来减少出版偏见。然而,还是存在一定的局限性:我们的审查仅考虑完全发表的研究,未发表的研究和会议摘要不在分析范围内,因为所需数据不可用;我们的搜索仅限于以英文或中文发表的研究,因为作者和读者通常无法访问其他语言;此外,由于数据不足,四项研究被排除在外,而在这排除的四项研究中,GPER表达与BC生存率之间没有显著相关性。
尽管我们进行了全面分析,但仍有一些限制待解决:首先,为了最小化异质性,纳入的研究需要通过IHC检测GPER表达,这是最常用的方法。一方面,IHC的方法差异可能导致异质性。另一方面,第一抗体对IHC的敏感度有显著影响。一系列用于检测蛋白质的抗体稀释程度也有所不同。其他因素如储存时间和暴露时间,也可能导致潜在的偏差。由于纳入的文献中许多具体数据资料不全,不足以对异质性来源通过亚组分析的方法进行探索,因此本文通过采用随机效应模型进行合并的方法来处理异质性,这也是文章的局限之处。其次,在BC患者的GPER评估中,没有一致的阈值来定义阳性表达。作为生物标志物,预测值也可能随着阳性的不同数量/扩展而波动。在不同实验室使用不同抗体的不同染色方案中,免疫反应性可能不同,这也是本文存在的不足。最后,需要解释HR评估的方法。对于不直接报告HR和95%CI的研究,根据已发表文章中提到的可用数据进行计算。即使没有可用的数据,我们也必须根据公布的方法从生存曲线中推断出这个值。这种方法可能会由于读取生存曲线的不准确而导致错误,因此我们尝试通过两个评估者独立读取曲线来最小化错误。估计的HR似乎不如直接获得的HR可靠。因此,我们将估计的HR和95%CI与公布的结果进行了比较,以确保估计的HR的准确性,严格阐述分析结果和结论。
未来需要进一步的研究来阐明GPER在乳腺癌中的相关分子机制和调控网络,以制定预防、诊断和治疗策略。
Competing interests: The authors declare that they have no competing interests.作者贡献:赵俞乔:查阅文献及撰写论文刘广麟、王海存、高欣:指导论文姜兴明:论文选题及思路设计刘浪:论文修改 -
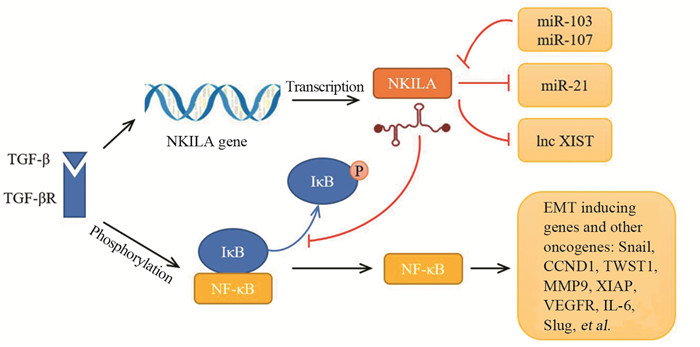
表 1 NKILA在肿瘤中的功能作用
Table 1 Function of NKILA in cancers

-
[1] Mattick JS. Non-coding RNAs: the architects of eukaryotic complexity[J]. EMBO Rep, 2001, 2(11): 986-991. doi: 10.1093/embo-reports/kve230
[2] 王微娜, 毕永红, 张瑞芹, 等. 消化系统恶性肿瘤中BANCR的表达及调控作用研究[J]. 肿瘤学杂志, 2020, 26(2): 145-149. https://www.cnki.com.cn/Article/CJFDTOTAL-XHON202002014.htm Wang WN, Bi YH, Zhang RQ, et al. Expression and Regulatory Effects of BANCR in Digestive System Malignancies[J]. Zhong Liu Xue Za Zhi, 2020, 26(2): 145-149. https://www.cnki.com.cn/Article/CJFDTOTAL-XHON202002014.htm
[3] 栗浩然, 王雅莉, 李红娟, 等. lncRNA FAL1通过调控MAPK通路对卵巢癌细胞化疗耐药性的影响及其机制[J]. 肿瘤防治研究, 2021, 48(4): 333-340. doi: 10.3971/j.issn.1000-8578.2021.20.0752 Li HR, Wang YL, Li HJ, et al. Effect of lncRNA FAL1 on Chemotherapy Resistance of Ovarian Cancer Cells by Regulating MAPK Pathway and Related Mechanism[J]. Zhong Liu Fang Zhi Yan Jiu, 2021, 48(4): 333-340. doi: 10.3971/j.issn.1000-8578.2021.20.0752
[4] Xue H, Yu P, Wang WZ, et al. The reduced lncRNA NKILA inhibited proliferation and promoted apoptosis of chondrocytes via miR-145/SP1/NF-κB signaling in human osteoarthritis[J]. Eur Rev Med Pharmacol Sci, 2020, 24(2): 535-548.
[5] Li QY, Li PR, Su JL, et al. LncRNA NKILA was upregulated in diabetic cardiomyopathy with early prediction values[J]. Exp Ther Med, 2019, 18(2): 1221-1225. https://www.ncbi.nlm.nih.gov/pubmed/31316617
[6] Yang YM, Li YH, Ding LL, et al. Regulatory effect of lncRNA NKILA on autophagy induced by sepsis kidney injury[J]. Eur Rev Med Pharmacol Sci, 2020, 24(8): 4057. https://pubmed.ncbi.nlm.nih.gov/31599426/
[7] Liu Q, Liu Z, Zhou LJ, et al. The long noncoding RNA NKILA protects against myocardial ischaemic injury by enhancing myocardin expression via suppressing the NF-κB signalling pathway[J]. Exp Cell Res, 2020, 387(2): 111774. doi: 10.1016/j.yexcr.2019.111774
[8] Liu BD, Sun LJ, Liu Q, et al. A cytoplasmic NF-κB interacting long noncoding RNA blocks IκB phosphorylation and suppresses breast cancer metastasis[J]. Cancer Cell, 2015, 27(3): 370-381. doi: 10.1016/j.ccell.2015.02.004
[9] Luo LH, Rao L, Luo LF, et al. Long non-coding RNA NKILA inhibited angiogenesis of breast cancer through NF-κB/IL-6 signaling pathway[J]. Microvasc Res, 2020, 129: 103968. doi: 10.1016/j.mvr.2019.103968
[10] Li DM, Zhong M, Su QB, et al. Active fraction of Polyrhachisvicina Rogers (AFPR) suppressed breast cancer growth and progression via regulating EGR1/lncRNA-NKILA/NF-κB axis[J]. Biomed Pharmacother, 2020, 123: 109616. doi: 10.1016/j.biopha.2019.109616
[11] Duan SZ, Chan WK, Oman A, et al. NF-κB/NKILA signaling modulates the anti-cancerous effects of EZH2 inhibition[J]. J Cell Mol Med, 2019, 23(9): 6182-6192. doi: 10.1111/jcmm.14500
[12] Wu W, Chen F, Cui XY, et al. LncRNA NKILA suppresses TGF-β-induced epithelial-mesenchymal transition by blocking NF-κB signaling in breast cancer[J]. Int J Cancer, 2018, 143(9): 2213-2224. doi: 10.1002/ijc.31605
[13] Tibatan MA, ÖnayUçar E. Long non-coding RNA NKILA regulates expression of HSP90α, NF-κB and β-catenin proteins in the MCF-7 breast cancer cell line[J]. Mol Biol Rep, 2021, 48(5): 4563-4571. doi: 10.1007/s11033-021-06482-y
[14] Wang FP, Jiang XC, Wang P. NF-κB interaction long non-coding RNA inhibits migration, invasion and epithelial-mesenchymal transition of cervical cancer cells through inhibiting NF-κB signaling pathways[J]. Exp Ther Med, 2020, 20(2): 1039-1047. doi: 10.3892/etm.2020.8752
[15] Wang J, Zhu Z, Qiu H, et al. LncRNA NKILA inhibits the proliferation and promotes the apoptosis of CSCC cells by downregulating miRNA-21[J]. J Cell Physiol, 2020, 235(11): 7863-7869. doi: 10.1002/jcp.29439
[16] 宁佳羽, 包伟晶, 周素娟, 等. LncRNA-MEG3和KLF4在鼻咽癌发生发展中的作用及可能机制[J]. 肿瘤防治研究, 2021, 48(3): 234-238. doi: 10.3971/j.issn.1000-8578.2021.20.0790 Ning JY, Bao WJ, Zhou SJ, et al. Role and Possible Mechanism of LncRNA-MEG3 and KLF4 in Carcinogenesis and Progression of Nasopharyngeal Carcinoma[J]. Zhong Liu Fang Zhi Yan Jiu, 2021, 48(3): 234-238. doi: 10.3971/j.issn.1000-8578.2021.20.0790
[17] Zhang W, Guo QN, Liu GY, et al. NKILA represses nasopharyngeal carcinoma carcinogenesis and metastasis by NF-κB pathway inhibition[J]. PLoS Genet, 2019, 15(8): e1008325. doi: 10.1371/journal.pgen.1008325
[18] Yang T, Li SS, Liu JJ, et al. lncRNA-NKILA/NF-κB feedback loop modulates laryngeal cancer cell proliferation, invasion, and radioresistance[J]. Cancer Med, 2018, 7(5): 2048-2063. doi: 10.1002/cam4.1405
[19] 席婕, 戴皓, 刘晖, 等. lncRNA-NKILA靶向NF-κB调控喉癌细胞的增殖和侵袭[J]. 医学分子生物学杂志, 2018, 15(6): 379-383. doi: 10.3870/j.issn.1672-8009.2018.06.004 Xi J, Dai H, Liu H, et al. LncRNA-NKILA Targets NF-κB Regulates Proliferation and Invasion of Laryngeal Carcinoma Cells[J]. Yi Xue Fen Zi Sheng Wu Xue Za Zhi, 2018, 15(6): 379-383. doi: 10.3870/j.issn.1672-8009.2018.06.004
[20] Lu ZL, Li Y, Wang JN, et al. Long non-coding RNA NKILA inhibits migration and invasion of non-small cell lung cancer via NF-κB/Snail pathway[J]. J Exp Clin Cancer Res, 2017, 36(1): 54. doi: 10.1186/s13046-017-0518-0
[21] Liu DM, Shi XY. Long non-coding RNA NKILA inhibits proliferation and migration of lung cancer via IL-11/STAT3 signaling[J]. Int J Clin Exp Pathol, 2019, 12(7): 2595-2603. https://www.ncbi.nlm.nih.gov/pubmed/31934087
[22] 胡增涛, 王微娜, 孙东升, 等. 食管鳞状细胞癌中环状RNA的生物学功能与调控机制研究[J]. 临床误诊误治, 2020, 33(3): 121-124. doi: 10.3969/j.issn.1002-3429.2020.03.027 Hu ZT, Wang WN, Sun DS, et al. Research on the biological function and regulatory mechanism of cyclic RNA in esophageal squamous cell carcinoma[J]. Lin Chuang Wu Zhen Wu Zhi, 2020, 33(3): 121-124. doi: 10.3969/j.issn.1002-3429.2020.03.027
[23] Lu ZL, Chen ZL, Li Y, et al. TGF-β-induced NKILA inhibits ESCC cell migration and invasion through NF-κB/MMP14 signaling[J]. J Mol Med (Berl), 2018, 96(3-4): 301-313. doi: 10.1007/s00109-018-1621-1
[24] Ke S, Li RC, Meng FK, et al. NKILA inhibits NF-κB signaling and suppresses tumor metastasis[J]. Aging (Albany NY), 2018, 10(1): 56-71. https://pubmed.ncbi.nlm.nih.gov/29348395/
[25] Tao F, Xu YP, Yang DZ, et al. LncRNA NKILA correlates with the malignant status and serves as a tumor-suppressive role in rectal cancer[J]. J Cell Biochem, 2018, 119(12): 9809-9816. doi: 10.1002/jcb.27300
[26] Jiang P, Han XT, Zheng YN, et al. Long non-coding RNA NKILA serves as a biomarker in the early diagnosis and prognosis of patients with colorectal cancer[J]. Oncol Lett, 2019, 18(2): 2109-2117. https://pubmed.ncbi.nlm.nih.gov/31423284/
[27] Chen RG, Cheng QY, Owusu-Ansah KG, et al. NKILA, a prognostic indicator, inhibits tumor metastasis by suppressing NF-κB/Slug mediated epithelial-mesenchymal transition in hepatocellular carcinoma[J]. Int J Biol Sci, 2020, 16(3): 495-503. doi: 10.7150/ijbs.39582
[28] Yu XL, Tang W, Yang YC, et al. Long noncoding RNA NKILA enhances the anti-cancer effects of baicalein in hepatocellular carcinoma via the regulation of NF-κB signaling[J]. Chem Biol Interact, 2018, 285: 48-58. doi: 10.1016/j.cbi.2018.02.027
[29] Zhang GD, Li Y, Liao GJ, et al. LncRNA NKILA inhibits invasion and migration of osteosarcoma cells via NF-κB/Snail signaling pathway[J]. Eur Rev Med Pharmacol Sci, 2019, 23(10): 4118-4125. https://pubmed.ncbi.nlm.nih.gov/31173281/
[30] Bian DH, Gao C, Bao K, et al. The long non-coding RNA NKILA inhibits the invasion-metastasis cascade of malignant melanoma via the regulation of NF-κB[J]. Am J Cancer Res, 2017, 7(1): 28-40. https://pubmed.ncbi.nlm.nih.gov/28123845/
[31] Lyu XM, Ma YQ, Wu F, et al. LncRNA NKILA inhibits retinoblastoma by downregulating lncRNA XIST[J]. Curr Eye Res, 2019, 44(9): 975-979. doi: 10.1080/02713683.2019.1606253
[32] Huang W, Cui XY, Chen JN, et al. Long non-coding RNA NKILA inhibits migration and invasion of tongue squamous cell carcinoma cells via suppressing epithelial-mesenchymal transition[J]. Oncotarget, 2016, 7(38): 62520-62532. doi: 10.18632/oncotarget.11528
[33] Hu DY, Zhong T, Dai Q. Long Non-Coding RNA NKILA Reduces Oral Squamous Cell Carcinoma Development Through the NF-KappaB Signaling Pathway[J]. Technol Cancer Res Treat, 2020, 19: 1533033820960747. https://pubmed.ncbi.nlm.nih.gov/33143574/
[34] Chen Z, Li ST, Shen L, et al. NF-kappa B interacting long noncoding RNA enhances the Warburg effect and angiogenesis and is associated with decreased survival of patients with gliomas[J]. Cell Death Dis, 2020, 11(5): 323. doi: 10.1038/s41419-020-2520-2
-
期刊类型引用(5)
1. 胡雨荷, 臧云彩, 李姗, 王颖睿, 申昕, 孙婷婷, 郑玉玲. 整合医学下基于圆运动及一气周流理论论治喉癌. 中医学报. 2025(08)  百度学术
百度学术
2. 刘川,马玮,王志海,李彦仕,潘敏,曾泉,胡国华. pT3N0期喉鳞癌的临床治疗策略. 临床耳鼻咽喉头颈外科杂志. 2025(01): 61-65 .  百度学术
百度学术
3. 彭丽娜,武川军,要兆旭,赵倩,韩海平. 沉默miR-373对喉癌细胞增殖、凋亡能力的影响及作用机制研究. 中国耳鼻咽喉头颈外科. 2024(06): 346-350 .  百度学术
百度学术
4. 张寒,张胜利,祖媛媛. 声门上型喉癌手术患者预后的预测模型构建. 国际医药卫生导报. 2024(11): 1796-1801 .  百度学术
百度学术
5. 郭珊珊,杨文婧,许丽萍,陶倩,王欢,王书谦. 喉癌治疗后复发的影响因素分析及预测模型构建. 山东医药. 2023(33): 27-31 .  百度学术
百度学术
其他类型引用(0)




 下载:
下载: