Expression of Four Long Non-coding RNAs in Breast Cancer and Their Relation with Patient's Prognosis
-
摘要:目的
探讨EGOT、MEG3、KCNQ1OT1和NEAT1四种LncRNA在不同亚型乳腺癌患者中的表达及与患者临床病理特征及预后的关系。
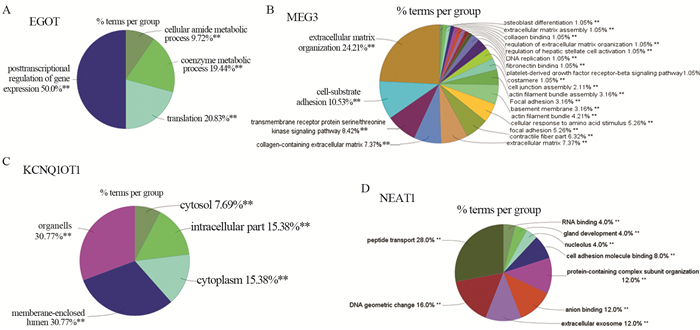
方法利用UALCAN网站挖掘TCGA数据库等大数据,分析四种LncRNA在1 097个乳腺癌组织样本和114个正常组织样本中的表达,用Kaplan-Meier Plotter法绘制生存曲线,分析四种LncRNA与乳腺癌患者预后的关系。通过LinkedOmics网站和ClueGO工具分析与它们表达相关的蛋白质的主要功能。
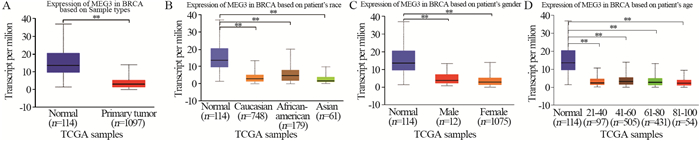
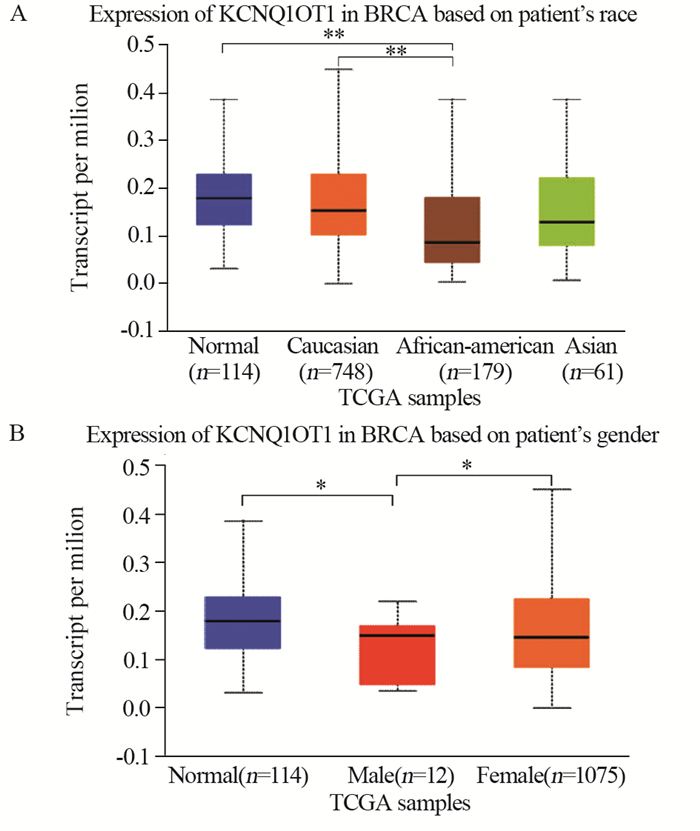
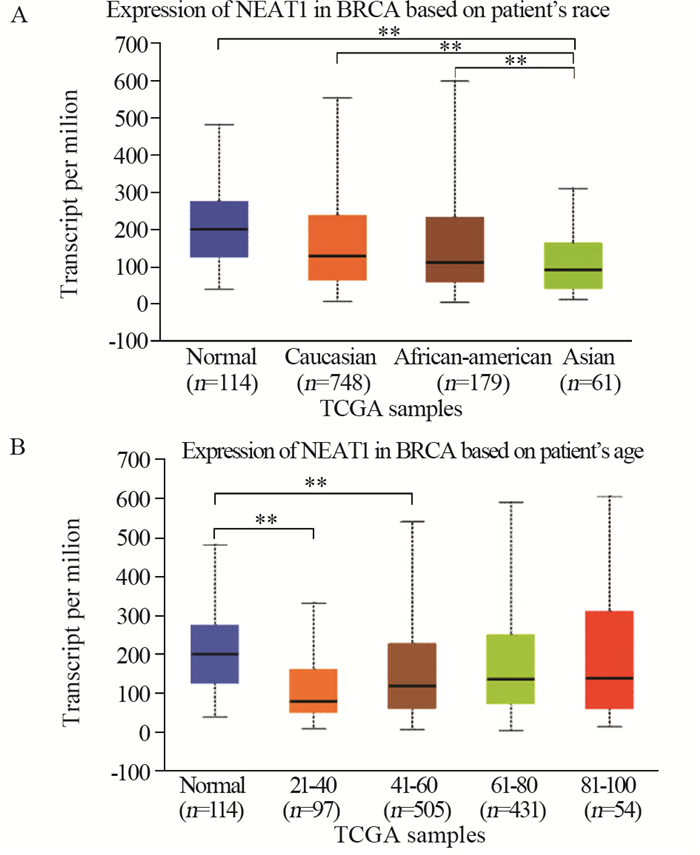
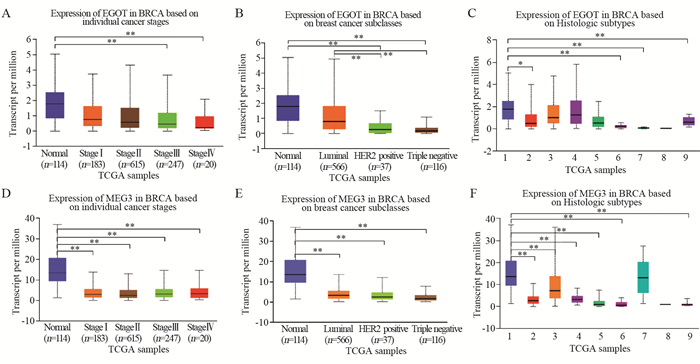
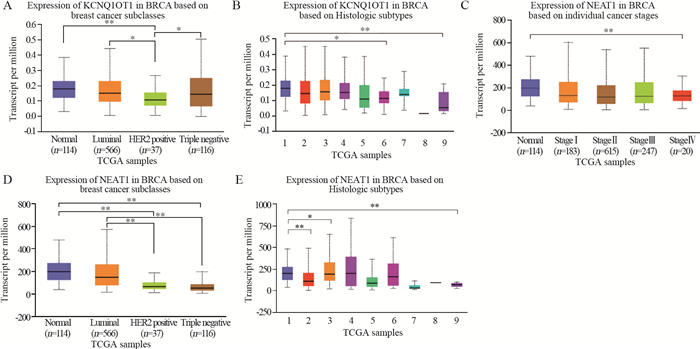
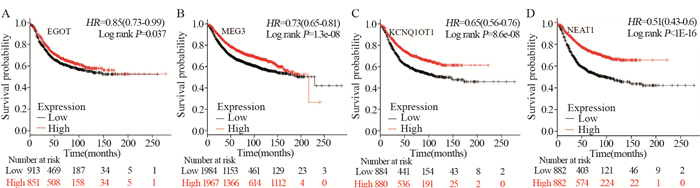
结果EGOT、MEG3在乳腺癌组织中的表达显著低于正常组织(P < 0.05);HER2阳性乳腺癌中KCNQ1OT1显著低表达(P < 0.05);NEAT1在Ⅳ期、HER2阳性和三阴性乳腺癌中的表达显著低于正常组织(P < 0.05)。四种LncRNA的表达水平与乳腺癌患者的无复发生存时间(RFS)均有显著相关性(P < 0.05),且LncRNA低表达时乳腺癌患者的RFS和预后较差。通过功能富集分析发现与四种LncRNA表达相关的蛋白质均与重要生物过程或通路相关。
结论四种LncRNA在乳腺癌或部分亚型中表达下调,它们可能是乳腺癌中潜在的抑癌因子,且可成为判断乳腺癌患者预后的生物学指标。
Abstract:ObjectiveTo investigate the expression of EGOT, MEG3, KCNQ1OT1 and NEAT1 in breast cancer tissues and their correlation with clinicopathologic features and prognosis of breast cancer patients.
MethodsExpression of four lncRNAs in 1097 breast cancer samples and 114 normal samples in the TCGA database were analyzed using UALCAN website. Survival curves were described by Kaplan-Meier Plotter website to analyze the relation between lncRNAs and the prognosis of breast cancer patients. The main functions of the proteins related to their expression were analyzed by LinkedOmics and ClueGO tools.
ResultsThe expression of EGOT and MEG3 in breast cancer tissues were significantly lower than those in normal tissues (P < 0.05). Expression of KCNQ1OT1 was significantly lower in HER2-positive breast cancer (P < 0.05). The NEAT1 expression in stage Ⅳ breast cancer, HER2-positive and triple negative breast cancer tissues were significantly lower than that in normal tissues (P < 0.05). The expression levels of the four lncRNAs were significantly correlated with the relapse-free survival time (RFS) of breast cancer patients (P < 0.05), and the RFS and prognosis of breast cancer patients were relatively poor when lncRNA expression was low. The functional enrichment analysis results showed that all the proteins related to the expression of four lncRNAs were related to important biological processes or pathways.
ConclusionThe four lncRNAs (EGOT, MEG3, KCNQ1OT1 and NEAT1) are down-regulated in breast cancer or some subtypes. They may be potential tumor suppressor factors in breast cancer and biological indicators for the prognosis of breast cancer patients.
-
Key words:
- Long non-coding RNA /
- Breast cancer /
- Prognosis
-
0 引言
结直肠癌已成为威胁我国人民生命健康的重大疾病之一。基于世界卫生组织的最新统计数据,2020年我国结直肠癌新发病例55.5万,死亡病例38.6万,位居癌症新发病例的第二位、死亡病例的第四位,且呈现持续上升的趋势,防控形势严峻[1]。遵循全直肠系膜切除理念的外科手术治疗显著降低了直肠癌的局部复发率[2]。然而,吻合口漏依然是结直肠手术后常见的并发症之一,不仅增加住院时间与费用,也会影响患者的长期生存[3]。目前认为,回肠袢式造口通过肠内容物转流可以预防吻合口漏的发生或减轻肠吻合口漏的临床症状。对于吻合口漏发生风险较高的患者,往往行预防性回肠袢式造口[2, 4]。然而,回肠袢式造口影响患者生活质量,加重患者的躯体痛苦与心理负担[5]。因此,及时、安全的还纳显得尤为重要。
虽然造口还纳手术操作相对简单,技术难度较小,但受限于手术视野与术中粘连等因素,术后并发症仍难以完全避免。其中,小肠梗阻最为常见,其次为伤口感染,此外还有吻合口漏、吻合口出血、肠瘘、切口疝等[6-7]。这些并发症增加了患者住院费用,延长了住院时间,也加重了出院后护理负担[8]。因此,如何最大程度地减少还纳手术后并发症,已经为越来越多结直肠外科医生所关注。随着微创技术的发展,腹腔镜手术因其术野清晰、手术创伤小、术后恢复快与并发症少等优点,逐渐展示了在造口还纳手术中的应用前景。本文就腹腔镜技术在造口还纳手术中的研究进展进行分析,以期为其临床应用提供新思路。
1 腹腔镜技术在造口还纳手术中的优势
1.1 腹腔镜辅助的回肠袢式造口还纳
1971年,Turnbull等首先描述了回肠袢式造口技术,他们将其应用在中毒性降结肠的外科治疗中[9]。回肠袢式造口的还纳通常开腹进行,将小肠自切口提出,体外裁剪、裸化肠管后完成消化道重建。开腹行还纳手术往往会造成小肠在吻合口处或腹腔内的粘连,而这些粘连是导致术后小肠梗阻的重要原因[10-11]。基于这个理论,Royds等设计了一项随机对照试验(randomized controlled trial, RCT)。在他们的研究中,共34名患者接受了传统开腹造口还纳手术,而另外40名患者同期行腹腔镜下腔内粘连分离,结果发现,接受了腹腔镜粘连分解的患者具有较短的住院时间(4天vs. 5天,P=0.003)和较少的住院花费(3 450欧元vs. 4 527欧元,P=0.015)[12]。更为重要的是,行腹腔镜辅助造口还纳手术的患者出现2例术后伤口感染、1例术后肺部感染与1例术后小肠梗阻,而对照组出现的术后并发症还包括肠梗阻(n=2)、肠外瘘(n=1)、肾积水(n=1)与尿潴留(n=3)。这些结果提示了腹腔镜辅助造口还纳手术具有较低的并发症发病率(10% vs. 23%, P=0.023)。此外,腹壁切口疝是还纳手术另一种常见并发症,腹腔镜下可以更直观地探查造口周围可能存在的腹壁缺损,也更容易使用补片进行加固。Young等的一项单中心回顾性研究发现,腹腔镜造口还纳手术中,肠粘连分解(60.4% vs. 26.3%, P < 0.01)与造口旁加固(32.1% vs. 6.3%, P < 0.01)机会要显著高于开腹手术,这也在一定程度上预防了腹壁切口疝的发生[13]。我国学者Wan等将腹腔镜辅助的造口还纳手术应用在克罗恩病的治疗中[14],发现其短期疗效也并不劣于开放手术,且存在更多肠粘连分解的机会(87.5% vs. 6.3%, P < 0.06)。考虑到造口周围肠管与腹壁、腹膜粘连紧密,开腹手术往往局部视野不清晰,腹腔镜可行腹腔内探查与操作的特点使其在造口还纳手术中具有得天独厚的优势。
1.2 腹腔镜辅助的Hartmann还纳
Hartmann手术由Henri Albert Hartmann于1923年提出,用于治疗结直肠肿瘤、复杂性憩室炎、左结肠穿孔或梗阻、缺血性结肠炎并发粪便污染水肿等疾病[15]。Hartman还纳手术通常开腹进行,但因腹腔内组织粘连较重、直肠残端显露困难等因素,开腹Hartman还纳手术具有10%的并发症发生率与4%的死亡率[16-17]。考虑到腹腔镜技术可以显著减少术后并发症,越来越多的外科医生将其应用在Hartman还纳术中。然而,迄今为止仍未有针对Hartman还纳手术的RCT研究。基于23例回顾性研究的meta分析显示,腹腔镜Hartman还纳手术显著减少了术后并发症(OR=0.53, 95%CI=0.47~0.58, P < 0.00001)。术后吻合口漏(OR=0.61, 95%CI=0.49~0.75, P < 0.00001)、腹腔感染(OR=0.67, 95%CI=0.52~0.87, P=0.003)、切口感染(OR=0.53, 95%CI=0.46~0.61, P < 0.00001)与肠梗阻(OR=0.46, 95%CI=0.29~0.72, P=0.0008)的风险也较开腹还纳手术显著降低[18]。需要指出的是,对于一些盆腔粘连较重、需要行切口疝修补等其他同期手术的患者来说,腹腔镜Hartman还纳术往往很难开展[19];既往Hartman手术为急诊开腹手术致使组织粘连重、肠道炎性反应较重的患者往往也不适合行腹腔镜还纳[20]。而这种选择偏倚是否会扩大腹腔镜Hartman还纳术的治疗效果仍需进一步的RCT研究来明确。
2 腹腔镜造口还纳手术中吻合方式的选择
2.1 吻合器吻合较手工吻合的优势
随着吻合器在上世纪80年代的发明,越来越多的肠管吻合使用吻合器进行。对于造口还纳手术而言,吻合器与手工吻合均可采用[21]。然而,有研究认为,手工吻合可能使吻合口变窄,术后的水肿也会进一步加重吻合口的狭窄[22]。因此,更多学者使用器械吻合以减少还纳手术后肠梗阻的发生。最新纳入4 917例行造口还纳手术患者的荟萃分析显示[23],吻合器吻合较体外吻合而言,显著降低了术后肠梗阻发生率(5.58% vs. 7.03%, OR=0.69, 95%CI=0.51~0.92, P=0.01)而不增加吻合口漏的发生率(2.08% vs. 2.93%,OR=0.81, 95%CI=0.34~1.54, P=0.52)。此外,多项研究显示,吻合器吻合可显著缩短造口还纳手术时间,患者术后胃肠功能恢复更快,术后住院日降低[24-25]。尽管吻合器吻合可能增加手术费用,但这些费用可以被缩短的手术与术后住院时间所抵消[26]。
2.2 体内吻合技术在造口还纳中的应用
腹腔镜的广泛应用与吻合器械的进步使体内消化道重建成为可能,从而可以实现完全腹腔镜手术。完全腹腔镜手术中腹腔脏器暴露在空气中的时间短,对腹腔组织及微环境影响也较小,可最大程度上促进胃肠道功能的恢复。目前,针对完全腹腔镜下右半结肠癌的RCT研究已经证实了体内吻合较体外吻合胃肠功能恢复快(2.3天vs. 3.3天,P=0.003),术后肠梗阻发生率也有所降低(13% vs. 30%,P=0.022)[27]。近年来,越来越多的学者将腹腔镜下体内吻合技术应用于回肠造口还纳手术,并取得了满意的近期疗效。Sujatha-Bhaskar等回顾性收集了132例行回肠袢式造口的患者资料[2],其中,49例患者于腹腔镜下完成肠粘连分解后行体外吻合,33例患者于腹腔镜下使用线性切割闭合器行肠管侧侧吻合,50例行开腹造口还纳手术。他们发现较体外吻合与开腹还纳手术而言,腹腔镜下体内吻合术后平均住院时间缩短(52.1 h vs. 69.6 h vs. 69 h, P < 0.05),但术后并发症发生率(6.1% vs. 14.3% vs. 10%, P > 0.05)与总住院费用(10 761美元vs. 11 274美元vs. 10 386美元,P > 0.05)没有明显增加。2019年,日本学者Kita等将线性切割闭合器自体外插入回肠造口,先完成近端与远端肠管的吻合、再于腹腔镜下置入切割闭合器关闭肠管共同开口,对9例患者实现了完全腹腔镜下的造口还纳手术[28]。较传统开腹手术而言,这种手术方式也并未增加患者术后并发症发生率(22.2% vs. 50%, P=0.64)与住院时间(11天vs. 12天,P=0.39)。
值得一提的是,开腹造口还纳需经皮肤切除造口并将肠管提出腹腔以完成消化道重建,而肥胖患者因腹壁肥厚,往往需要更大的手术切口,进而带来了更高的切口感染风险。因此,完全腹腔镜下造口还纳可能在肥胖患者中更具有应用优势。自2018年起,笔者团队开展了完全腹腔镜下造口还纳手术:先于腹腔镜下使用切割闭合器离断造口远端与近端肠管,再行两段肠管对系膜侧重叠式侧侧吻合,后使用另一切割闭合器关闭共同开口完成体内消化道重建。之后,逐层进腹切除造口、一期缝合。我们对比了30例完全腹腔镜造口还纳与34例开腹还纳在肥胖患者(BMI > 28 kg/m2)中的手术效果[29],结果显示行完全腹腔镜造口还纳手术的患者手术切口更短(6.5 cm vs. 8.5 cm, P < 0.001),术后排气更快(2天vs. 3天,P=0.017),切口感染风险更低(6.7% vs. 26.5%, P=0.049)。相较于完全腹腔镜造口还纳,开腹还纳手术还出现了肠梗阻(n=1, 2.94%)、吻合口漏(n=1, 2.94%)与腹腔出血(n=1, 2.94%)等并发症。这些研究结果从不同角度说明了完全腹腔镜技术在回肠造口还纳手术中的应用潜力,但其作用仍需要多中心、大样本的前瞻性RCT研究来进一步明确。
3 总结与展望
综上所述,使用腹腔镜行造口还纳手术,一方面可使手术视野更加清晰,另一方面更有利于腹腔粘连分解、腹壁缺损修补与腔内消化道重建等操作。应用腹腔镜行还纳手术在不显著增加手术费用的前提下,减少了手术创伤,加快了术后胃肠功能恢复,降低了术后并发症。不过,这些优势还需要更多RCT研究证实。此外,目前的研究并未严格规范腹腔镜还纳手术的适应症,是否所有拟行还纳手术的患者均适合应用这一技术也值得进一步探索;由于造口的存在与腔内消化道重建增加了感染风险,手术中更应严格遵循无菌原则,对于刚开始从事腹腔镜手术与腔内吻合的医生来说也存在一定的技术挑战。但毋庸置疑的是,现有的研究已为该技术在临床进一步推广提供了一部分循证医学证据,相信未来腹腔镜造口还纳手术会在临床获得更加广泛的应用。
作者贡献苏嘉庆:实验设计、实施及论文撰写李智伟:实验设计、实验指导万召娟、王芳华:实验实施汪梦、张恒阳、张慧欣:实验数据分析周百灵:实验设计、论文审校 -
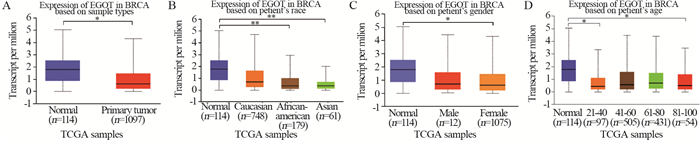
图 5 EGOT和MEG3在不同临床病理特征乳腺癌患者中的表达
*: P < 0.05; **: P < 0.01. 1: Normal(n=114); 2: IDC(n=784); 3: ILC(n=203); 4: Mixed(n=29); 5: Other(n=45); 6: Mucinous(n=17); 7: Metaplastic(n=9); 8: INOS(n=1); 9: Medullary(n=6).
Figure 5 Expression of EGOT and MEG3 in breast cancer patients with different clinicopathological characteristics
图 6 KCNQ1OT1和NEAT1在不同临床病理特征乳腺癌患者中的表达
*:P < 0.05;**:P < 0.01;HER2:human epidermal growth factor receptor-2;IDC:infiltrating ductal carcinoma; ILC: infiltrating lobular carcinoma; Mixed: mixed histology; Mucinous: mucinous carcinoma; Medullary: medullary carcinoma; Metaplastic: metaplastic carcinoma; INOS: infltrating carcinoma no otherwise specifed.
Figure 6 Expression of KCNQ1OT1 and NEAT1 in breast cancer patients with different clinicopathological characteristics
表 1 与LncRNA表达相关的蛋白质
Table 1 Proteins related to expression of four lncRNAs

-
[1] Miller KD, Nogueira L, Mariotto AB, et al. Cancer treatment and survivorship statistics, 2019[J]. CA Cancer J Clin, 2019, 69(5): 363-385. doi: 10.3322/caac.21565
[2] 卢振东, 孙晓晓, 谢龙祥, 等.乳腺癌分子分型研究进展[J].河南大学学报(医学版), 2018, 37(4): 229-233. http://d.old.wanfangdata.com.cn/Periodical/zgsyzdx201106079 Lu ZD, Sun XX, Xie LX, et al. The development of molecular subtyping in breast cancer[J]. Henan Da Xue Xue Bao (Yi Xue Ban), 2018, 37(4): 229-233. http://d.old.wanfangdata.com.cn/Periodical/zgsyzdx201106079
[3] Yuan CL, Jiang XM, Yi Y, et al. Identification of differentially expressed lncRNAs and mRNAs in luminal-B breast cancer by RNA-sequencing[J]. BMC cancer, 2019, 19(1): 1-12. doi: 10.1186/s12885-018-5219-3
[4] 皮亚平, 刘黎明. BCYRN1对乳腺癌细胞MCF7和小鼠移植瘤增殖和转移的影响[J].肿瘤防治研究, 2018, (4): 205-209. doi: 10.3971/j.issn.1000-8578.2018.17.0691 Pi YP, Liu LM. Influence of BCYRN1 on proliferation and migration of breast cancer cell line MCF7 and tumor-bearing mice[J]. Zhong Liu Fang Zhi Yan Jiu, 2018, (4): 205-209. doi: 10.3971/j.issn.1000-8578.2018.17.0691
[5] Rhodes DR, Kalyana-Sundaram S, Mahavisno V, et al. Oncomine 3.0: genes, pathways, and networks in a collection of 18, 000 cancer gene expression profiles[J]. Neoplasia, 2007, 9(2): 166-180. doi: 10.1593/neo.07112
[6] Tomczak K, Czerwinska P, Wiznerowicz M. The Cancer Genome Atlas (TCGA): an immeasurable source of knowledge[J]. Contemp Oncol (Pozn), 2015, 19(1A): A68-A77. http://d.old.wanfangdata.com.cn/Periodical/zhcmj201918010
[7] Zhou B, Zhao H, Yu J, et al. EVLncRNAs: a manually curated database for long non-coding RNAs validated by low-throughput experiments[J]. Nucleic Acids Res, 2018, 46(D1): D100-D105. doi: 10.1093/nar/gkx677
[8] Gao Y, Wang P, Wang Y, et al. Lnc2Cancer v2.0: updated database of experimentally supported long non-coding RNAs in human cancers[J]. Nucleic Acids Res, 2019, 47(D1): D1028-D1033. doi: 10.1093/nar/gky1096
[9] Chandrashekar DS, Bashel B, Balasubramanya SAH, et al. UALCAN: A Portal for Facilitating Tumor Subgroup Gene Expression and Survival Analyses[J]. Neoplasia, 2017, 19(8): 649-658. doi: 10.1016/j.neo.2017.05.002
[10] Lánczky A, Nagy Á, Bottai G, et al. miRpower: a web-tool to validate survival-associated miRNAs utilizing expression data from 2178 breast cancer patients[J]. Breast Cancer Res Treat , 2016, 160(3): 439-446. http://cn.bing.com/academic/profile?id=bef9b13ef201039040bc8d00ccc58220&encoded=0&v=paper_preview&mkt=zh-cn
[11] Vasaikar SV, Straub P, Wang J, et al. LinkedOmics: analyzing multi-omics data within and across 32 cancer types[J]. Nucleic Acids Res, 2018, 46(D1): D956-D963. doi: 10.1093/nar/gkx1090
[12] Xu SP, Zhang JF, Sui SY, et al. Downregulation of the long noncoding RNA EGOT correlates with malignant status and poor prognosis in breast cancer[J]. Tumor Biol, 2015, 36(12): 9807-9812. doi: 10.1007/s13277-015-3746-y
[13] Jin L, Quan J, Pan X, et al. Identification of lncRNA EGOT as a tumor suppressor in renal cell carcinoma[J]. Mol Med Rep, 2017, 16(5): 7072-7079. doi: 10.3892/mmr.2017.7470
[14] 陈梦雪, 李学良.长链非编码RNA在食管癌中的研究进展[J].肿瘤防治研究, 2018, 45(11): 932-935. doi: 10.3971/j.issn.1000-8578.2018.18.0399 Chen MX, Li XL. Research Progress of Long Non-coding RNA in Esophageal Cancer[J]. Zhong Liu Fang Zhi Yan Ju, 2018, 45(11): 932-935. doi: 10.3971/j.issn.1000-8578.2018.18.0399
[15] Ghafouri-Fard S, Taheri M. Maternally expressed gene 3 (MEG3): A tumor suppressor long non coding RNA[J]. Biomed Pharmacother, 2019, 118: 109129. doi: 10.1016/j.biopha.2019.109129
[16] Moradifard S, Hoseinbeyki M, Ganji SM, et al. Analysis of microRNA and gene expression profiles in Alzheimer's disease: a meta-analysis approach[J]. Sci Rep, 2018, 8(1): 4767. http://cn.bing.com/academic/profile?id=02dd3edaf2a89a92f51c98e259cb56c7&encoded=0&v=paper_preview&mkt=zh-cn
[17] Feng W, Wang C, Liang C, et al. The dysregulated expression of KCNQ1OT1 and its interaction with downstream factors miR-145/CCNE2 in breast cancer cells[J]. Cell Physiol Biochem, 2018, 49: 432-446. doi: 10.1159/000492978
[18] Shin VY, Chen J, Cheuk IW, et al. Long non-coding RNA NEAT1 confers oncogenic role in triple-negative breast cancer through modulating chemoresistance and cancer stemness[J]. Cell Death Dis, 2019, 10(4): 270. doi: 10.1038/s41419-019-1513-5



 下载:
下载: