文章信息
- 白璇,唐慧,郭强.
- BAI Xuan, TANG Hui, GUO Qiang.
- 大肠癌细胞FoxQ1与EGFR基因表达的相关性
- Relationship Between FoxQ1 and Epidermal Growth Factor Receptor Gene Expression in Colorectal Cancer
- 肿瘤防治研究, 2016, 43(01): 20-24
- Cancer Research on Prevention and Treatment, 2016, 43(01): 20-24
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2016.01.005
-
文章历史
- 收稿日期: 2014-12-20
- 修回日期: 2015-04-20
2. 650032 昆明,云南省第一人民医院(昆明医科大学附属昆华医院)临床基础医学研究所
2. Institute of Basic Medical Research, The First People’s Hospital of Yunnan Province(Affiliated Kunhua Hospital of Kunming Medical University), Kunming 650032, China
叉头框(forkhead box,Fox)蛋白家族是一类转录因子家族,其DNA结合区具有一个高度保守的翼状螺旋结构。根据DNA结合区的同源性,目前将该蛋白家族分为19个亚群(从FoxA到FoxS)[1, 2],FOX蛋白作为转录因子调控诸多下游基因的表达,参与包括细胞周期、增殖、分化、凋亡、衰老及其肿瘤的浸润及转移等生物学进程,与细胞命运的转归和肿瘤的发生发展息息相关[3]。表皮生长因子受体(epidermal growth factor receptor,EGFR)是一广泛分布于人体各组织细胞膜上的多功能糖蛋白,是鸟类成红细胞白血病病毒(avian erythroblastic leukemia viral,v-erb-b)致癌基因同源体,EGFR基因的过度表达能促进正常细胞的转化和肿瘤细胞的转移,EGFR信号通路在肿瘤的形成和发展过程中占据重要地位[4]。本研究组在前期工作中构建了基于shRNA慢病毒干扰系统的FOXQ1基因沉默DLD1大肠癌细胞株[5],本研究中应用该细胞株就FoxQ1与EGFR基因的相关性进行了初步探讨,为进一步研究FoxQ1基因在EGFR信号通路中的作用及分子机制奠定实验基础。
1 材料和方法 1.1 材料EGFR酪氨酸激酶抑制剂盐酸埃罗替尼(Erlotinib HCl)购自Selleck公司(美国);EGFR siRNA购自Santa Cruz公司(美国);大肠癌细胞系DLD1、HT29、LOVO、HCT116和人胚肾细胞293-T均购自中国科学院细胞库;RPMI 1640培养液及0.05%胰蛋白酶均购自Hyclone公司(美国);澳洲特级胎牛血清及青霉素-链霉素溶液购自BioInd公司(以色列);Lipofectamine2000购自Invitrogen公司(美国);Biozol购自Biomiga公司(美国);GoScontroliptTM Reverse Transcontroliption System试剂盒购自Promega公司(美国);SYBR Premix Ex TaqⅡ购自TaKaRa公司(日本);6孔细胞培养板及25 cm2细胞培养皿购自BD Falcon公司(美国);RNA浓度测定所用仪器为NanoDrop(美国);实时定量PCR扩增仪为罗氏LightCycler 480(瑞士)。
1.2 方法 1.2.1 设计合成RT-PCR引物根据FOXQ1基因序列设计引物,上游引物FOXQ1-F:5’-GGCAACGGGCTACAGCTTTA-3’,下游引物FOXQ1-R:5’-GGCACCCCACATACATAATCAA-3’。EGFR基因上游引物EGFR-F:5’-ATGCTCTACAACCCCACCAC-3’,下游引物EGFR-R:5’-GCCCTTCGCACTTCTTACAC-3’。GAPDH作为内参基因,上游引物GAPDH-F:5’-TGACTTCAACAGCGACACCCA-3’,下游引物GAPDH-R:5’-CACCCTGTTGCTGTAGCCAAA-3’。引物由上海生工生物工程有限公司合成。
1.2.2 分别培养大肠癌细胞系DLD1、HT29、HCT116、LOVO和人胚肾细胞293-T分别接种大肠癌细胞DLD1、HT29、HCT116、LOVO和人胚肾细胞293-T于6孔板中,每孔3×105个细胞,用含10%胎牛血清的RPMI 1640完全培养液于5%CO2、37℃培养箱培养48 h。
1.2.3 以不同浓度的EGFR酪氨酸激酶抑制剂Erlotinib HCl处理DLD1细胞(经Erlotinib HCl处理的细胞以“EGFR-inhibitor/”开头命名)接种DLD1细胞于6孔板中,每孔3.5×105个细胞,用含10%胎牛血清的RPMI 1640培养液(不含双抗)于37℃、5%CO2培养箱培养24 h后,分别加入终浓度为30、50 μg/ml的Erlotinib HCl继续培养DLD1细胞72 h。 实验分组为:DLD1细胞;终浓度为30 μg/ml的Erlotinib HCl处理的DLD1细胞(EGFR-inhibitor-30/DLD1);终浓度为50 μg/ml的Erlotinib HCl处理的DLD1细胞(EGFR-inhibitor-50/DLD1)。
1.2.4 siRNA-EGFR转染DLD1-shRNA-FoxQ1细胞(经siRNA-EGFR转染的细胞以“siRNA-EGFR/”开头命名)接种DLD1-shRNA-FoxQ1细胞于6孔板,每孔3.5×105个细胞,用含10%胎牛血清的RPMI 1640培养液(不含双抗)于37℃、5%CO2培养箱培养24 h后,以2 μg/5 μl siRNA-EGFR/Lipofectamine 2000转染各组细胞6 h后弃去转染上清液,重新加入含10%胎牛血清的RPMI 1640完全培养液继续培养48 h。实验所用细胞:①:DLD1-shRNA-Control为不含siRNA-FOXQ1序列的空载体慢病毒干扰的DLD1细胞;②:DLD1-shRNA-FoxQ1为经shRNA-FoxQ1慢病毒干扰的DLD1细胞。实验分组:对照组:①和②细胞;siRNA-EGFR转染组:经siRNA-EGFR转染的①细胞(siRNA-EGFR/DLD1-shRNA-Control)和经siRNA-EGFR转染的②细胞(siRNA-EGFR/DLD1-shRNA-FoxQ1)。
1.2.5 EGFR酪氨酸激酶抑制剂Erlotinib HCl处理DLD1-shRNA-FoxQ1细胞接种DLD1-shRNA-FoxQ1细胞于6孔板,每孔3.5×105个细胞,用含10%胎牛血清的RPMI 1640培养液(不含双抗)于37℃、5%CO2培养箱培养24 h后,以终浓度为50 μg/ml的Erlotinib HCl处理各组细胞,用含10%胎牛血清的RPMI 1640完全培养液继续培养72 h。实验所用①和②细胞同1.2.4。实验分组为:对照组:①和②细胞;Erlotinib HCl处理组:经Erlotinib HCl处理的①细胞(EGFR-inhibitor/DLD1-shRNA-Control)和经Erlotinib HCl处理的②细胞(EGFR-inhibitor/DLD1-shRNA-FoxQ1)。
1.2.6 荧光定量PCR检测各组细胞FoxQ1及EGFR基因相对表达量用Biozol裂解各组细胞,提取细胞总RNA,取2 μg RNA经Promega反转录试剂盒反转录获得cDNA,取2 μl cDNA,应用SYBR Green及实时定量PCR扩增仪扩增FOXQ1及EGFR基因,收集荧光信号,用2-ΔΔCt法计算FOXQ1及EGFR基因在各组细胞中的相对表达量。
1.3 统计学方法实验所得数据采用(x±s)表示,各组之间的比较采用单因素方差分析(ANOVA)进行组间差异显著性检验,以P<0.05为差异用统计学意义。
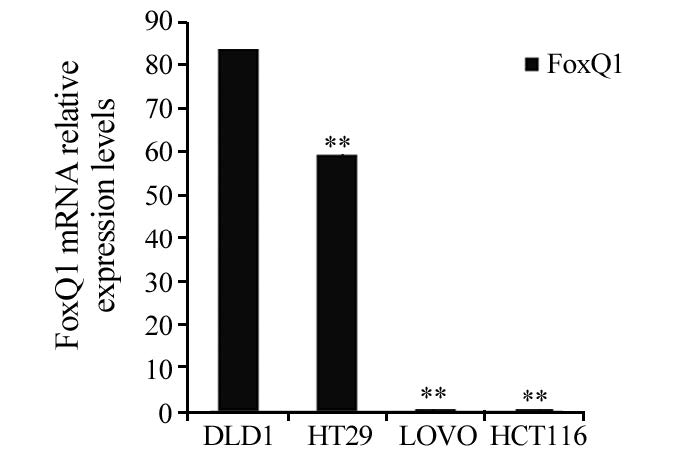
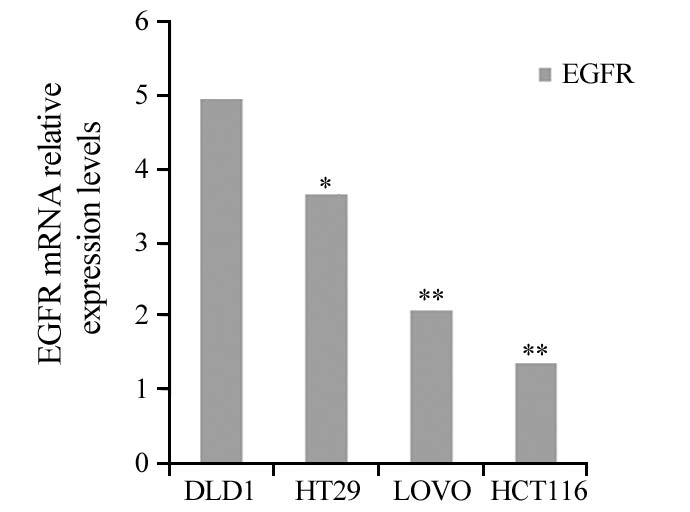
2 结果 2.1 大肠癌细胞系中FoxQ1及EGFR基因的相对表达量荧光定量PCR检测结果显示,以293-T细胞中FoxQ1相对表达量为1作为对照,FoxQ1在DLD1、HT29、LOVO、HCT116中的相对表达量分别为83.09、59.58、0.06、0.03,FoxQ1在DLD1中的相对表达量最高,其次是HT29,而在HCT116和LOVO中的相对表达量极低(DLD1-HT29:P=0.009,DLD1-LOVO:P=0.000,DLD1-HCT116:P=0.000),见图 1。 以293-T细胞中EGFR相对表达量为1作为对照 ,EGFR在DLD1、 HT29、LOVO、HCT116中的相对表达量分别为4.95、3.67、2.08、1.36,EGFR在DLD1中的相对表达量最高,其次是HT29,在LOVO和HCT116中也有表达,但显著低于DLD1和HT29(DLD1-HT29:P=0.028,DLD1-LOVO:P=0.003,DLD1-HCT116:P=0.000),见图 2。大肠癌细胞系中FoxQ1与EGFR基因的表达趋势基本一致。
|
| **: P<0.01, compared with DLD1 cells 图 1 FoxQ1 mRNA在大肠癌细胞系中的相对表达量 Figure 1 FoxQ1 mRNA relative expression in colorectal cancer cell line |
|
| *: P<0.05, **: P<0.01, compared with DLD1 cells 图 2 EGFR mRNA在大肠癌细胞系中的相对表达量 Fig. 2 Epidermal growth factor receptor (EGFR) mRNA relative expression in colorectal cancer cell line |
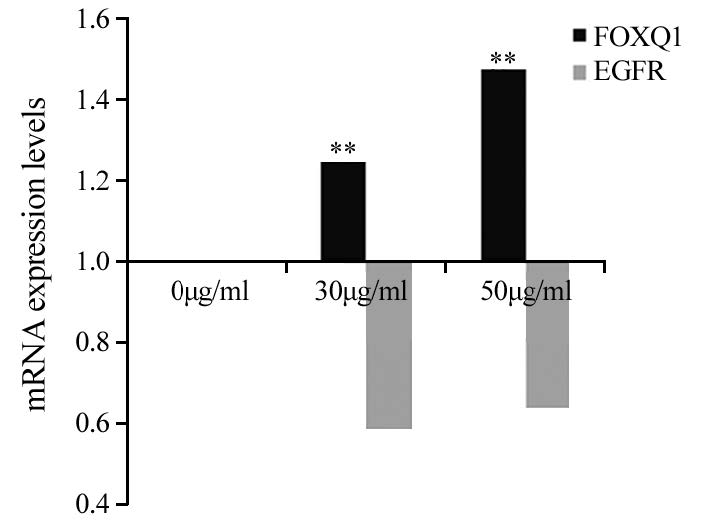
荧光定量PCR检测结果显示,DLD1细胞经不同浓度Erlotinib HCl处理阻断EGFR酪氨酸激酶后,EGFR表达量降低,但在Erlotinib HCl浓度为30和50 μg/ml时差异无统计学意义;而FoxQ1的表达量经Erlotinib HC处理后均升高,且在Erlotinib HCl浓度为50 μg/ml时显著高于30 μg/ml(P=0.000),见图 3。
|
| **: P<0.01, pairwise compared with DLD1 cells 图 3 不同浓度EGFR酪氨酸激酶抑制剂Erlotinib HCl处理的DLD1细胞中FoxQ1及EGFR基因mRNA相对表达量 Fig. 3 FoxQ1 and EGFR mRNA relative expression in DLD1 cells treated with different concentrations of Erlotinib HCl |
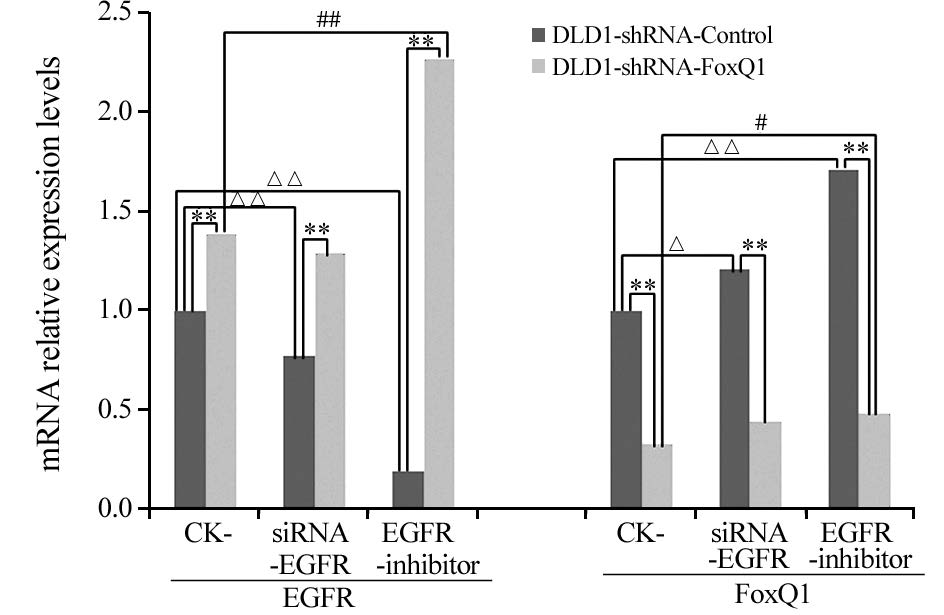
荧光定量PCR结果显示:(1)DLD1-shRNA-Control和DLD1-shRNA-FoxQ1组间比较:在空白对照组、siRNA-EGFR和EGFR酪氨酸激酶抑制剂Erlotinib HCl处理组中,FoxQ1的表达在DLD1-shRNA-FoxQ1细胞较DLD1-shRNA-Control细胞均极显著降低(P=0.002、0.001、0.002),而EGFR表达在DLD1-shRNA-FoxQ1细胞较DLD1-shRNA-Control细胞均极显著增高(P=0.000、0.002、0.002)。(2)DLD1-shRNA-Control组内比较: DLD1-shRNA-Control细胞经siRNA-EGFR和EGFR酪氨酸激酶抑制剂Erlotinib HCl处理组分别与空白对照组相比,EGFR表达量均极显著降低(P=0.003、0.002),而FoxQ1基因表达量均显著增高(P=0.043、0.002)。(3)DLD1-shRNA-FoxQ1组内比较:DLD1-shRNA-FoxQ1细胞经siRNA-EGFR处理后,EGFR表达量的降低与空白对照组相比无统计学意义,FoxQ1的升高与空白对照组相比亦无统计学意义;但EGFR酪氨酸激酶抑制剂Erlotinib HCl处理组与空白对照组相比,EGFR表达量反而极显著升高(P=0.007),FoxQ1基因的表达量也显著升高(P=0.041),见图 4。
|
| **: P<0.01: compared between DLD1-shRNA-Control and DLD1-shRNA-FoxQ1 groups: Δ: P<0.05, ΔΔ: P<0.01: compared within DLD1-shRNA-Control groups; #: P<0.05, ##: P<0.01: compared within DLD1-shRNA-FoxQ1 groups 图 4 FoxQ1 mRNA在各处理组细胞中的相对表达量 Fig. 4 FoxQ1 mRNA relative expression in each treatment group |
人FoxQ1基因定位于人体6号染色体短臂25.3区(6p25.3),全长2 319 bp,仅含1个外显子,编码403个氨基酸,其功能可以稳定结合靶基因启动子区域的GC盒等核心元件,控制基因转录活性而发挥生物效应[6]。 FoxQ1作为叉头框蛋白转录因子家族中的一员,生物学功能涉及毛囊形成和胃上皮的分化[7],参与胃酸分泌和黏蛋白基因的表达[8]。FoxQ1转录因子能结合到E-钙黏蛋白(E-cadherin,ECD)基因启动子区的E-box并抑制其表达,诱导上皮间质转化(epithelial mesenchymal transition,EMT)[9]。近年研究发现FoxQ1基因在人类肿瘤包括大肠癌、肺癌、乳腺癌和肝癌中异常表达 ,并促进肿瘤转移 [10, 11, 12, 13]。
人EGFR基因位于第7号染色体p13~q22区,全长200 kb,由28个外显子组成,编码1 186个氨基酸,其糖蛋白相对分子质量约170 kDa[14],与HER2/ErbB-2/Neu/p185、HER3/ErbB-3、HER4/ErbB-4等被归入HER/ErbB家族,同属于受体酪氨酸激酶(receptor tyrosine kinase,RTKs)。其配体通过与EGFR胞外结构特异性结合,激活EGFR的酪氨酸激酶活性,通过催化与肌醇磷代谢有关的酶、激酶、调节蛋白磷酸化,促进产生第二信使,导致核内原癌基因c-fos、c-myc表达,刺激肿瘤细胞生长增殖、黏附、侵袭和转移[15]。 EGFR通路近膜信号经过细胞质中各种衔接蛋白、酶等的级联反应到达核内,最主要的信号通路包括MAPK通路、PI3K通路或c-Src通路。Akt是PI3K下游一个重要的靶分子,通过使大量蛋白(包括调节FasL表达、Forkhead家族转录因子、caspase-9、GSK-3β、NF-κB)的磷酸化来调控细胞的存活和凋亡[16]。如Akt磷酸化FoxO的丝氨酸/苏氨酸残基,磷酸化的FoxO与DNA的亲和力下降,并从细胞核转移到细胞质,与细胞质中的伴侣蛋白14-3-3蛋白结合,阻止FoxO逆转运至细胞核内,从而抑制FoxO转录活性,Akt活性降低时,FoxO发生去磷酸化,FoxO进入细胞核内,转录活性被激活,并结合靶基因对应的DNA靶向序列,行使其转录功能[17, 18]。
然而,FoxQ1在肿瘤组织中的异常表达与EGFR-PI3K-Akt通路之间是否存在相关性,目前尚无文献报道。在本研究中,发现FoxQ1和EGFR基因的表达趋势在多个大肠癌细胞系中趋于一致,但是将DLD1细胞的FoxQ1基因敲低后,EGFR的表达反而升高。为了解释这一现象,进一步运用RNA干扰和化学抑制剂,分别在转录水平和酪氨酸激酶水平上对EGFR的表达及其蛋白的活化进行干扰。
在进行DLD1-shRNA-Control和DLD1-shRNA-FoxQ1组间比较、DLD1-shRNA-Control组内比较时,发现这样一种现象,当FoxQ1的表达被敲低后,EGFR的表达升高;EGFR受siRNA干扰及其酪氨酸激酶活性被抑制后,FoxQ1的表达均升高。这一结果提示,在大肠癌中FoxQ1与EGFR基因之间可能存在相互负反馈调节机制,当FoxQ1表达被抑制后,EGFR-FoxQ1轴所涉及的某些基因会被激活或抑制,从而负反馈调节EGFR基因,促使其表达升高;反之,在EGFR的表达或蛋白激酶活性被抑制后,EGFR-FoxQ1轴所涉及的某些基因被激活或抑制,从而促使FoxQ1表达升高。并且,在DLD1-shRNA-FoxQ1组内比较时,EGFR酪氨酸激酶抑制剂Erlotinib HCl处理组与空白对照组相比,EGFR表达量显著升高,FoxQ1基因的表达量也显著升高,提示虽然DLD1-shRNA-FoxQ1细胞中EGFR蛋白活性被抑制,但因FoxQ1表达被敲低,EGFR mRNA表达水平与DLD1-shRNA-FoxQ1细胞空白对照组相比反而明显升高,但因DLD1-shRNA-FoxQ1细胞中EGFR蛋白活性被抑制,FoxQ1 mRNA的表达水平与DLD1-shRNA-FoxQ1细胞空白对照组相比有所升高。此现象进一步验证了EGFR与FoxQ1基因之间存在相互负反馈调控机制的可能性。
EGFR-FoxQ1轴所涉及的基因是哪些,这些基因又是通过何种分子机制调控大肠癌细胞中EGFR及FoxQ1基因的表达,维持两者的高表达状态,并最终促进大肠癌的发生发展,这些问题将是我们今后的研究重点。
| [1] | Myatt SS, Lam EW. The emerging roles of forkhead box (Fox) proteins in cancer[J]. Nature Rev Cancer, 2007, 7(11): 847-59. |
| [2] | Kaestner KH, Knochel W, Martinez DE. Unified nomenclature for the winged helix/forkhead transcription factors[J]. Genes Dev, 2000, 14(2): 142-6. |
| [3] | Lam EW, Brosens JJ, Gomes AR, et al. Forkhead box proteins: tuning forks for transcriptional harmony[J]. Nat Rev Cancer, 2013: 13(7): 482-95. |
| [4] | Wu JH, Xie QL, Chen XJ, et al. Epidermal growth factor receptor (EGFR) and its signal transduction[J]. Sheng Ming Ke Xue, 2006, 18(2): 116-22. [吴健虹, 谢秋玲, 陈小佳, 等. 表皮生长因子受体EGFR及其信号传导[J]. 生命科学, 2006, 18(2): 116-22.] |
| [5] | Bai X, Tang H, Lang FC, et al. Construction of lentivirus expression vector and to silence the expression of FOXQ1 gene in colorectal cancer cell line DLD-1[J]. Shi Jie Hua Ren Xiao Hua Za Zhi, 2014, 22(19): 2752-7. [白璇, 唐慧, 郎丰超, 等. 慢病毒表达载体的构建及沉默FoxQ1基因在大肠癌细胞系DLD-1中的表达[J]. 世界华人消化杂志, 2014, 22(19): 2752-7.] |
| [6] | Bieller A, Pasche B, Frank S, et al. Isolation and characterization of the human foxkhead gene FOXQ1[J]. DNA Cell Biol, 2001, 20(9): 555-61. |
| [7] | Feuerborn A, Srivastava PK, Küffer S, et al. The Forkhead factor FoxQ1 influences epithelial differentiation[J]. J Cell Physiol, 2011, 226(3): 710-9. |
| [8] | Verzi MP, Khan AH, Ito S, et al. Transcription factor foxq1 controls mucin gene expression and granule content in mouse stomach surface mucous cells[J]. Gastroenterology, 2008, 135(2): 591-600. |
| [9] | Zhang H, Meng F, Liu G, et al. Forkhead transcription factor foxq1 promotes epithelial-mesenchymal transition and breast cancer metastasis[J]. Cancer Res, 2011, 71(4): 1292-301. |
| [10] | Kaneda H, Arao T, Tanaka K, et al. FOXQ1 is overexpressed in colorectal cancer and enhances tumorigenicity and tumor growth[J]. Cancer Res, 2010, 70(5): 2053-63. |
| [11] | Qiao Y, Jiang X, Lee ST, et al. FOXQ1 regulates epithelial-mesenchymal transition in human cancers[J]. Cancer Res, 2011, 71(8): 3076-86. |
| [12] | Feng J, Zhang X, Zhu H, et al. FoxQ1 overexpression influences poor prognosis in non-small cell lung cancer, associates with the phenomenon of EMT[J]. PLoS One, 2012, 7(6): e39937. |
| [13] | Xia L, Huang W, Tian D, et al. Forkhead Box Q1 Promotes Hepatocellular Carcinoma Metastasis by Transactivating ZEB2 and VersicanV1 Expression[J]. Hepatology, 2014, 59(3): 958-73. |
| [14] | Reiter JL, Threadgill DW, Eley GD, et al. Comparative genomic sequence analysis and isolation of human and mouse alternative EGFR transcripts encoding truncated receptor isoforms[J]. Genomics, 2001, 71(1): 1-20. |
| [15] | Lei W, Mayotte JE, Levitt ML. Enhancement of chemosensitivity and PCD by tyrosine kinase inhibitors correlates with EGFR in NSCLC cells[J]. Anticancer Res, 1999, 19(1A): 221-8. |
| [16] | Ellert-Miklaszewska A, Kaminskab B, Konarska L. Cannabinoids down-regulate PI3K/Akt and Erk signalling pathways and activate proapoptotic function of Bad protein[J]. Cell Signal, 2005, 17(1): 25-37. |
| [17] | Fabre S, Lang V, Bismuth G, et al. PI3-kinase and the control of T cell growth and proliferation by FoxOs[J]. Bull Cancer, 2006, 93(5): E36-8. |
| [18] | Van Der Heide LP, Hoekman MF, Smidt MP, et al. The ins and outs of FoxO shuttling: mechanisms of FoxO translocation and transcriptional regulation[J]. Biochem, 2004, 380(Pt 2): 297-309. |
 2016, Vol. 43
2016, Vol. 43


