文章信息
- 刘晓光,陈兆峰,刘敏,贾娇,魏丽琼,叶玉伟,周永宁. 2015.
- LIU Xiaoguang, CHEN Zhaofeng, LIU Min, JIA Jiao, WEI Liqiong, YE Yuwei, ZHOU Yongning. 2015.
- 微小RNA-133b在胃癌中的表达及意义
- miR-133b Expression in Gastric Cancer and Related Significance
- 肿瘤防治研究, 2015, 42(05): 474-477
- Cancer Research on Prevention and Treatment, 2015, 42 (05): 474-477
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2015.05.011
-
文章历史
- 收稿日期:2014-06-06
- 修回日期:2014-11-27
胃癌是人类最常见的恶性肿瘤之一,在全球范围内,胃癌发病率居恶性肿瘤第4位,病死率居恶性肿瘤第2位[1]。癌细胞的侵袭及转移是造成死亡的主要原因[1, 2]。因此,探索胃癌浸润转移的机制,需找一种新的具有高敏感度和高特异性的肿瘤标志物对早期胃癌诊断、治疗及病情监测都具有显著意义。
微小RNA(microRNA,miRNA)是一类广泛存在于多种生物体内、序列长约19~24(see miRBase,http://microrna.sanger.ac.uk)个核苷酸、不编码蛋白质的单链RNA,其能够通过与对应的靶mRNA的3’非翻译区(3’UTR)的非完全或完全配对结合,从而阻止其翻译和(或)破坏其稳定性,实现其对基因表达的调控[3]。据研究,在哺乳动物中miRNAs可调控超过60%的蛋白编码基因[4],参与调控人类约30%的基因调控[5],控制着几乎所有细胞分化、增殖和程序性死亡、肿瘤发生等多种病理、生理过程[6]。 已有研究证实miRNAs在不同肿瘤中表达上调或下调,这一现象表明miRNAs在肿瘤的发生发展过程中发挥着重要的作用[7]。MiR-133b位于染色体6p12.2,最初被认为是一种心肌和骨骼肌特异性表达的microRNA,可调节肌肉发育及肌信号通路相关基因[8]。已有研究报道miR-133b在肺癌[9, 10]、膀胱癌[11]、食管癌[12]等多种肿瘤组织中呈低表达,表明其可能发挥抑癌基因的作用,与肿瘤的发生、发展密切相关。目前miR-133b在胃癌发生、发展中的作用研究甚少。为此,本实验应用miRNAs芯片对胃癌组织及癌旁正常组织的microRNA表达谱进行分析,并经RT-PCR验证,为进一步研究miR-133b调控胃癌发生、发展的具体信号通路奠定基础。
1 材料与方法 1.1 材料7种人胃癌细胞株:HGC-27(未分化)、BGC-803(未分化)、MKN-45(低分化)、MGC-823(低分化)、SGC-7901(中分化)、NCL-N87(高分化)、AGS(高分化)及正常胃黏膜上皮永生化细胞株GES-1均购自中国科学院上海生命科学研究院。RPMI1640及DMEM(Dulbecco modified Ealge’s medium)培养液购自美国Gibco公司,细胞培养用新生牛血清购自杭州四季清公司,胰蛋白酶购自美国Sigma公司,RNAisoTM Plus(日本TaKaRa公司,Code:D9108A)、One Step PrimeScript® miRNA Cdna Synthesis Kit(日本TaKaRa公司,Code:D350A),SYBP® Premix Ex TaqTM Ⅱ(日本TaKaRa公司,Code:DRR081A),Universal miRNA qPCR Control Primer Set(日本TaKaRa公司,Code:D356),has-miR-133b qPCR Primer Mix (日本TaKaRa公司,Code:DHM0097)。
1.2 方法 1.2.1 细胞培养GES-1细胞株用含10%新生胎牛血清、青霉素(100 u/ml)和链霉素(100 μg/ml)的DMEM培养液培养,其余细胞均在含10%新生牛血清、青霉素(100 u/ml)和链霉素(100 μg/ml)的RPMI1640培养液中,37℃、5% CO2和95%空气的恒温培养箱中培养,细胞贴壁生长,2天换液一次,3~4天传代一次,待细胞长至80%~90%融合时,取处于对数生长期的细胞用于实验。
1.2.2 标本收集收集2009年10月—2010年4月甘肃省武威肿瘤医院接受手术治疗的56例胃癌患者作为研究对象。取胃癌及其相应癌旁正常组织(距离癌灶5 cm以上,且经HE染色证实均无癌细胞)进行研究,其中癌组织56例,相应癌旁正常组织56例,术中取材,立即放入液氮中保存后转入-80℃长期保存。分期以国际抗癌联盟(International Union Against Cancer,UICC)/美国肿瘤联合会(American Joint Committee on Cancer,AJCC)TNM病理分期第六版为标准。患者均有完整的临床资料,包括姓名、性别、年龄、肿瘤直径、肿瘤部位、分化程度、浸润深度、淋巴结转移、淋巴管侵犯和远处转移。56例患者中,男32例,女24例,男女比例为1.3:1,年龄35~76岁,中位年龄56岁。所有患者术前均未接受过放、化疗。
1.2.3 细胞与组织Small RNA提取按照RNAiso for Small RNA说明书提取总的Small RNA(20~200 nt) ,用紫外分光光度仪测定Small RNA的浓度,以及根据OD260/OD280判断Small RNA纯度。所有Small RNA OD260/OD280均在1.8~2.1之间,说明RNA纯度较好,可用于反转录反应(cDNA合成)。
1.2.4 反转录成cDNA 应用One Step PrimeScript®miRNA cDNA Synthesis Kit对提取的Small RNA进行Poly(A)加尾以及cDNA合成。首先按2×miRNA Reaction Buffer Mix 10 μl、0.1%BSA 2 μl、miRNA PrimeScript® RT Enzyme Mix 2 μl、总RNA(1 μg/μl)1 μl、RNase Free dH2O 5 μl,将以上反应液在冰上配制混合,在37℃ 60 min进行Pol(A)加尾和反转录反应,85℃ 5 s进行酶的失活反应。向所得到的RT反应液中添加80 μl RNase Free dH2O补足至100 μl用于Real-time PCR反应,将其置于-20℃保存。
1.2.5 miR-133b在人胃癌细胞株及胃癌组织表达的检测本实验用TaKaRa公司合成的has-miR-133b qPCR Primer Mix作为特异性引物,以U6 SnRNA作为内部参照。用LightCycler® Real-time PCR扩增仪进行miRNAs实时荧光定量RCR检测。每个样本重复3次。PCR反应以25 μl体系进行,包括1 μl经稀释的RT反应液,SYBP® Premix Ex TaqTM Ⅱ12.5 μl、has-miR-133b qPCR Primer Mix 1 μl、dH2O 10.5 μl,反应条件:95℃ 5 s,60℃ 20 s,循环40次。根据每孔荧光信号达到阈值时经历的循环数作为Ct值,以U6 SnRNA作为内参,使用相对定量法,用N=2-△△CT值表示miR-133b在胃癌细胞及组织中相对正常细胞及组织表达的倍数,此时△△CT=(CT肿瘤miR-133b-CT肿瘤U6)-(CT正常miR-133b-CT正常U6)。
1.3 统计学方法应用SPSS16.0统计软件包处理数据,计量资料结果以均数±标准差(x±s)表示,采用t检验进行两样本均数之间的比较,多个样本均数之间的比较采用单因素方差分析,P﹤0.05为差异有统计学意义。
2 结果 2.1 miR-133b在7种胃癌细胞株及正常胃黏膜细胞株中的表达 2.1.1 miR-133b在胃癌细胞中的表达通过miR-133b U6 SnRNA的熔解曲线可见real-time PCR产物的特异性较好,曲线为单峰,表明无引物二聚体扩增和非特异扩增,见图 1; 扩增曲线可见各样品的重复性好,各样品的扩增效率较一致,见图 2。
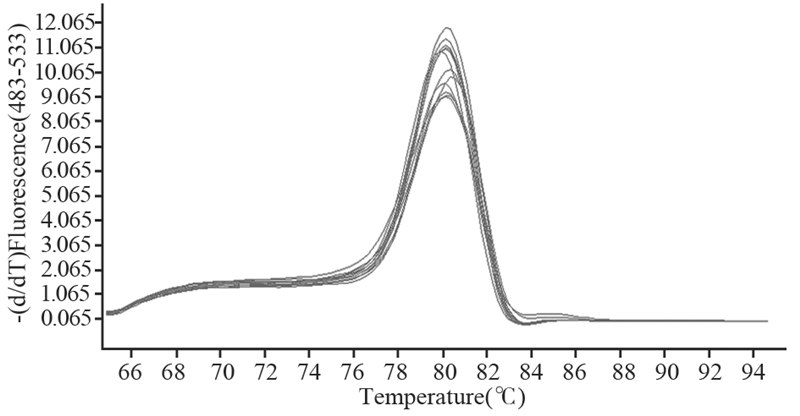
|
| 图 1 胃癌细胞中miR-133b的real-time PCR溶解曲线 Figure 1 miR-133b real-time PCR melting curves of gastric cancer cells |
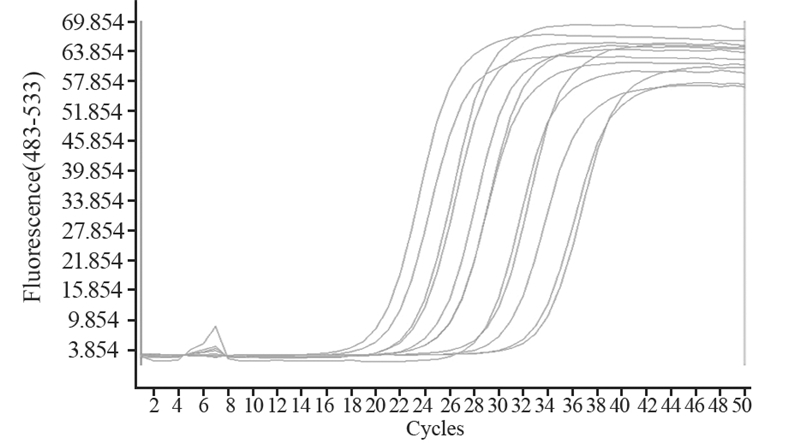
|
| explainnoteps 图 2 胃癌细胞中miR-133b的real-time PCR扩增曲线 Figure 2 miR-133b real-time PCR amplification curves of gastric cancer cells |
miR-133b在不同分化程度的7种胃癌细胞株中的表达均低于正常胃黏膜细胞株GES-1。miR-133b在HGC-27、MGC-803、MKN-45、BGC-823、SGC-7901、NCI-N87和AGS细胞株的表达量分别是0.0149±0.0192、0.0326±0.0315、0.0442±0.0324、0.0635±0.0432、0.0751±0.0598、0.1169±0.0313、0.4632±0.0792,其表达量与GES-1(1.0000)相比差异均有统计学意义(P < 0.05),且其表达量与胃癌细胞的分化程度呈正相关性,胃癌细胞的恶性度越差,其表达量越低。
2.2 miR-133b在胃癌组织中的表达及意义 2.2.1 miR-133b在胃癌及其癌旁组织中的表达以ΔCt值表示miR-133b在组织中的相对表达量。ΔCt值越高表明miR-133b表达越低。荧光定量PCR结果显示,miR-133b在胃癌组织中的ΔCt值为8.892±2.693,而癌旁正常组织的ΔCt值为6.188±1.563,表明miR-133b在胃癌组织中的表达低于癌旁正常组织,差异有统计学意义(P<0.01)。以N=2-△△CT表示肿瘤组织中miR-133b相对于癌旁正常组织表达倍数。N<1表示肿瘤组织miR-133b表达水平低于癌旁正常组织;N>1表示肿瘤组织中miR-133b的表达水平高于癌旁正常组织,56例中50例表达下调,其中42例表达下调≥2.0倍;6例表达上调,其中上调≥2倍有3例,见图 3。
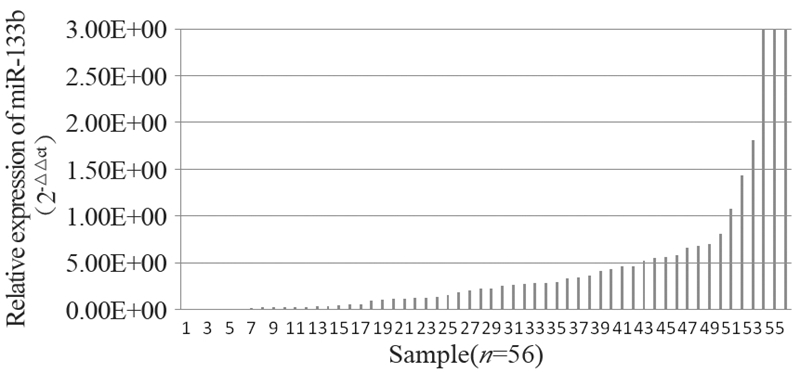
|
| explainnoteps 图 3 miR-133b在胃癌组织较相应癌旁组织的表达倍数(2-ΔΔCt) Figure 3 The expression of miR-133b compared between gastric cancer tissues and matched adjacent non-tumor tissues(2-ΔΔCt) |
对56例胃癌组织标本中miR-133b的表达水平与患者临床病理参数之间相关性进行统计分析发现,胃癌组织miR-133b表达与分化程度、TNM分期及有无淋巴结转移有关。与性别、年龄及肿瘤大小无关,见表 1。
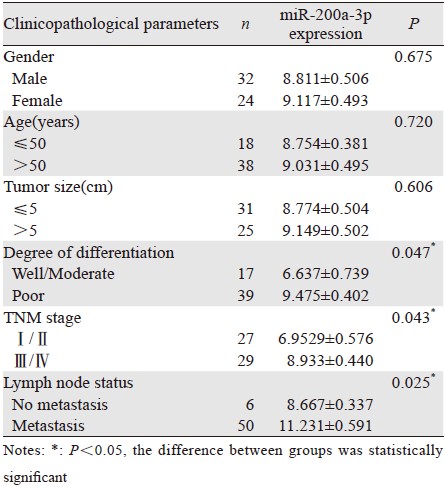 |
胃癌的治疗主要分为手术、化疗、放疗、免疫治疗和中医中药治疗等,其预后与病理分期、部位、组织类型、治疗措施等有关[13],即使行根治性手术复发率亦较高。胃癌的发生、发展过程与细胞异常增殖和抗凋亡密切相关,涉及基因及蛋白质层面众多肿瘤相关因子的异常表达及功能改变[14]。许多研究证实,通过调节不同靶基因的表达,miRNAs在不同的癌症中发挥癌基因或者抑癌基因的作用。Lim等[15]研究发现一个miRNA可以调控多个mRNA,并且多个miRNA可以调控一个mRNA。目前已发现let-7a[16]、miR-148a[17]、miR-152[17]、miR-551[18]及miR-200a/200b/429[19]等在胃癌中表达下调,导致其调控的促癌靶蛋白脱离控制,表达上调,致使肿瘤细胞增殖过度、凋亡缺失,从而导致肿瘤的发生、发展;另有研究证实miR-191、miR-107、miR-25、miR-221、miR-21[20, 21]等表达上调,发挥癌基因的作用,并证实与胃癌的发生、发展等生物学行为密切相关。因此,对miRNAs在胃癌中差异表达进行研究,对寻找胃癌诊断和治疗靶点有着深远意义。
miR-133b是心肌和骨骼肌特异表达的miRNA,其在心肌调控发面的研究较多。最近,研究发现其在多种肿瘤中表达下调,可能表现为抑癌基因的作用。Hu等[22]研究发现,miR-133b在结直肠癌细胞中的表达显著降低,并随着细胞恶性程度的增高而减低,可有效抑制肿瘤细胞的增殖、促进细胞凋亡,对细胞迁移及侵袭能力有抑制作用,并且证实EMT及MMP9可能是miR-133b作用的靶基因,miR-133b在转录后水平抑制EMT和MMP9的表达,发挥抑癌基因的作用。Kano等[12]研究证实,miR-133b在食管鳞状上皮细胞癌中通过作用于靶基因FSCN1发挥抑癌基因的作用。另有研究证实miR-133b在肺癌[9]、膀胱癌[23]、舌部鳞癌[24]等肿瘤中表达下调,转染miR-133b可抑制肿瘤细胞的增殖和侵袭能力,可能发挥抑癌基因的作用。
本试验表明miR-133b在胃癌细胞和组织中表达下调,提示其可能在胃癌的发生、发展中发挥抑癌基因的作用。本研究结果与Guo等[25]在胃癌中的研究一致。但miR-133b在胃癌差异表达的具体意义及其生物学功能尚不清楚。本试验为后续深入研究miR-133b在胃癌发生、发展中的生物学功能及其介导的分子机制奠定了基础,可进一步探讨胃癌中miR-133b与靶基因的相互作用。
| [1] | Crew KD, Neugut AI. Epidemiology of gastric cancer[J]. World J Gastroenterol, 2006, 12(3): 354-62. |
| [2] | Parkin DM, Bray F, Ferlay J, et al. Global cancer statistics, 2002[J]. CA Cancer J Clin, 2005, 55(2): 74-108. |
| [3] | Melo SA, Ropero S, Moutinho C, et al. A TARBP2 mutation in human cancer impairs microRNA processing and DICER1 function[J]. Nat Genet, 2009, 41(3): 365-70. |
| [4] | Friedman RC, Farh KK, Burge CB, et al. Most mammalian mRNAs are conserved targets of microRNAs[J]. Genome Res, 2009, 19(1): 92-105. |
| [5] | Filipowicz W, Bhattacharyya SN, Sonenberg N. Mechanisms of post-transcriptional regulation by microRNAs: are the answers in sight? [J]. Nat Rev Genet, 2008, 9(2): 102-14. |
| [6] | . Bartel DP. MicroRNAs: target recognition and regulatory functions[J]. Cell, 2009, 136(2): 215-33. |
| [7] | Calin GA, Croce CM. MicroRNA signatures in human cancers[J]. Nat Rev Cancer, 2006, 6(11): 857-66. |
| [8] | Xu C, Lu Y, Pan Z, et al. The muscle-specific microRNAs miR-1 and miR-133 produce opposing effects on apoptosis by targeting HSP60, HSP70 and caspase-9 in cardiomyocytes[J]. J Cell Sci, 2007, 120(Pt 17): 3045-52. |
| [9] | Crawford M, Batte K, Yu L, et al. MicroRNA 133B targets pro-survival molecules MCL-1 and BCL2L2 in lung cancer[J]. Biochem Biophys Res Commun, 2009, 388(3): 483-9. |
| [10] | Wu Y, Crawford M, Yu B, et al. MicroRNA delivery by cationic lipoplexes for lung cancer therapy[J]. Mol Pharm, 2011, 8(4): 1381-9. |
| [11] | Song T, Xia W, Shao N, et al. Differential miRNA expression profiles in bladder urothelial carcinomas[J]. Asian Pac J Cancer Prev, 2010, 11(4): 905-11. |
| [12] | Kano M, Seki N, Kikkawa N, et al. miR-145, miR-133a and miR-133b: Tumor-suppressive miRNAs target FSCN1 in esophageal squamous cell carcinoma[J]. Int J Cancer, 127(12): 2804-14. |
| [13] | Parkin DM, Pisani P, Ferlay J. Global cancer statistics[J]. CA Cancer J Clin, 1999, 49(1): 33-64, 1. |
| [14] | Hanahan D, Weinberg RA. The hallmarks of cancer[J]. Cell, 2000, 100(1): 57-70. |
| [15] | Lim LP, Lau NC, Garrett-Engele P, et al. Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs[J]. Nature, 2005, 433(7027): 769-73. |
| [16] | Motoyama K, Inoue H, Nakamura Y, et al.Clinical significance of high mobility group A2 in human gastric cancer and its relationship to let-7 microRNA family[J]. Clin Cancer Res, 2008, 14(8): 2334-40. |
| [17] | Chen Y, Song Y, Wang Z, et al. Altered expression of MiR-148a and MiR-152 in gastrointestinal cancers and its clinical significance[J]. J Gastrointest Surg, 2010, 14(7): 1170-9. |
| [18] | Li Z, Cao Y, Jie Z, et al. miR-495 and miR-551a inhibit the migration and invasion of human gastric cancer cells by directly interacting with PRL-3[J]. Cancer Lett, 2012, 323(1): 41-7. |
| [19] | Chang L, Huo BJ, Feng L, et al. Clinical significance and expression of miR-200a, miR-200b and miR-429 in gastric cancer[J]. Zhong Liu Fang Zhi Yan Jiu, 2013, 40(11): 1098-100. [常靓, 霍炳杰, 冯莉, 等. 胃癌组织中miR-200a, miR-200b和miR-429的表达及临床意义[J]. 肿瘤防治研究, 2013, 40(11): 1098-100.] |
| [20] | Volinia S, Calin GA, Liu CG, et al. A microRNA expression signature of human solid tumors defines cancer gene targets[J]. Proc Natl Acad Sci U S A, 2006, 103(7): 2257-61. |
| [21] | Zhang Z, Li Z, Gao C, et al. miR-21 plays a pivotal role in gastric cancer pathogenesis and progression[J]. Lab Invest, 2008, 88(12): 1358-66. |
| [22] | Hu G, Chen D, Li X, et al. miR-133b regulates the MET proto-oncogene and inhibits the growth of colorectal cancer cells in vitro and in vivo[J]. Cancer Biol Ther, 2010, 10(2): 190-7. |
| [23] | Dyrskj?t L, Ostenfeld MS, Bramsen JB, et al. Genomic profiling of microRNAs in bladder cancer: miR-129 is associated with poor outcome and promotes cell death in vitro[J]. Cancer Res, 2009, 69(11): 4851-60. |
| [24] | Wong TS, Liu XB, Chung-Wai Ho A, et al. Identification of pyruvate kinase type M2 as potential oncoprotein in squamous cell carcinoma of tongue through microRNA profiling[J]. Int J Cancer, 2008, 123(2): 251-7. |
| [25] | Guo J, Miao Y, Xiao B, et al. Differential expression of microRNA species in human gastric cancer versus non-tumorous tissues[J]. J Gastroenterol Hepatol, 2009, 24(4): 652-7. |
 2015, Vol. 42
2015, Vol. 42
